Classification of Mouse Transcription Factors
- Draft September 22, 2014 - |
| |
This classification is the working version that comprises only murine transcription factors (TFs) that have human orthologs, with few exceptions. In particular many mouse-specific TFs are still missing in Class 2.3 (C2H2 zinc finger factors). As for the classification of the human TFs, it may be subject to occasional changes, suggested for discussion.
Symbols / links:
 : TRANSFAC® class descriptions; : TRANSFAC® class descriptions;  : new class descriptions; : new class descriptions;  : additional information about the class and its members (see below); : additional information about the class and its members (see below);
 : Link to reviewed mouse entries UniProt; : Link to reviewed mouse entries UniProt;  : Link to unreviewed mouse entries UniProt; : Link to unreviewed mouse entries UniProt;  : Link to reviewed human UniProt entries; : Link to reviewed human UniProt entries;  / / : Link to reviewed/unreviewed rat UniProt entries, resp. : Link to reviewed/unreviewed rat UniProt entries, resp.
"Txxxxx": TRANSFAC accession numbers, mostly linked to BIOBASE Knowledge Library (BKL); "T" only provides a link to a BKL entry that has no TRANSFAC accession yet.
 : Link to PDB; preferably to a complex of the corresponding DNA-binding domain (DBD) with DNA, provisionally the DBD alone, referring to the human or a mammalian ortholog; for a few excpetions, illustrative structures of fungal homologs are linked. : Link to PDB; preferably to a complex of the corresponding DNA-binding domain (DBD) with DNA, provisionally the DBD alone, referring to the human or a mammalian ortholog; for a few excpetions, illustrative structures of fungal homologs are linked.
Additional information:
The question mark in the Class title line links to the class description, the sequences of the manually adjusted DNA-binding domains of this respective class and their alignments, logo plots and, whenever relevant, further information like dimerization matrices etc.
DNA sequence patterns:
The DNA sequences assigned to many families or subfamilies represent typical or proven individual binding sequences for (some of) the domains in the respective taxon. They should not be taken as a consensus, and they are not suitable for predicting potential TF binding sites.
The logo plots after many TF genera have been derived from the profile library published by the group of J. Taipale at Karolinska Institute (Jolma et al., Cell 152, 327-339 (2013); PubMed 23332764) and were kindly provided by P. Stegmaier (geneXplain GmbH). Each of these plots is linked to an htm file with all matrices for this factor, human and mouse, that were given by Jolma et al., one of them being selected for the logo plot. The individual matrices are given a unique ID, comprising the gene symbol of the factor and an alphanumeric specification of the respective matrix, which may be either a consecutive number, or may have an additional "m" to indicate monomeric factor binding mode, "d" for dimers, or "dd" for dimers of dimers bound to the respective sequence patterns. For further explanation of the information provided with each matrix, kindly refer to the original publication.
Citation:
Wingender, E., Schoeps, T. and Dönitz, J.: TFClass: An expandable hierarchical classification of human transcription factors. Nucleic Acids Res. 41, D165-D170 (2013). doi: 10.1093/nar/gks1123 (Link)
|
| |
| 1 |
Superclass: Basic domains |
 |
| |
1.1 |
Class: Basic leucine zipper factors (bZIP) |
 |
 |
| |
1.1.1 |
Family: Jun-related factors |
TGAGTCA |
|
| |
1.1.1.1 |
Subfamily: Jun factors |
TGAGTCA |
|
| |
1.1.1.1.1 |
c-Jun |
|
 |
T00131 |
 |
 |
 |
| |
1.1.1.1.2 |
JunB |
|
 |
T00436 |
 |
 |
|
| |
1.1.1.1.3 |
JunD |
|
 |
T00437 |
 |
 |
|
| |
1.1.1.2 |
Subfamily: NF-E2-like factors |
GCTGAGTCA |
|
| |
1.1.1.2.1 |
NF-E2 p45 |
 |
 |
T01441 |
 |
 |
|
| |
1.1.1.2.2 |
NF-E2L1 (NRF1) |
|
 |
T02203 |
 |
 |
|
| |
1.1.1.2.2.1 |
NF-E2L1, isoform 1 (Nrf1a) |
|
 |
T09558 |
|
| |
1.1.1.2.2.2 |
NF-E2L1, isoform 2 (Nrf1b) |
|
 |
T28874 |
|
| |
1.1.1.2.2.3 |
NF-E2L1, isoform 3 (short) |
|
 |
T13800 |
|
| |
1.1.1.2.3 |
NF-E2L2 (NRF2) |
|
 |
T04938 |
 |
 |
|
| |
1.1.1.2.4 |
NF-E2L3 (NRF3) |
|
 |
T05710 |
 |
 |
|
| |
1.1.1.2.5 |
BACH1 |
|
 |
T04793 |
 |
 |
|
| |
1.1.1.2.6 |
BACH2 |
|
 |
T04792 |
 |
 |
|
| |
1.1.1.3 |
Subfamily: ATF-2-like factors |
TGACGTCA |
|
| |
1.1.1.3.1 |
ATF-2 |
|
 |
T10238 |
 |
 |
 |
| |
1.1.1.3.1.1 |
ATF-2, isoform 1 |
|
 |
T02056 |
|
| |
1.1.1.3.1.2 |
ATF-2, isoform 2 |
|
 |
T01017 |
|
| |
1.1.1.3.1.3 |
ATF-2, isoform 3 |
|
 |
T02036 |
|
| |
1.1.1.3.2 |
ATF-7 |
 |
 |
T19847 |
 |
 |
|
| |
1.1.1.3.3 |
CREB-5 (CRE-BPa) |
 |
 |
T18881 |
 |
 |
|
| |
1.1.1.3.3.1 |
CREB-5, isoform 1 |
|
 |
T18880 |
|
| |
1.1.1.3.3.2 |
CREB-5, isoform 2 |
|
 |
T18882 |
|
| |
1.1.2 |
Family: Fos-related factors |
(TGAGTCA) |
|
| |
1.1.2.1 |
Subfamily: Fos factors |
(TGAGTCA) |
|
| |
1.1.2.1.1 |
c-Fos |
|
 |
T00122 |
 |
 |
 |
| |
1.1.2.1.2 |
FosB |
|
 |
T10574 |
 |
 |
|
| |
1.1.2.1.3 |
Fra-1 |
|
 |
T00292 |
 |
 |
|
| |
1.1.2.1.4 |
Fra-2 |
|
 |
T02199 |
 |
 |
|
| |
1.1.2.1.4.1 |
Fra-2, isoform 1 |
|
 |
T08535 |
|
| |
1.1.2.1.4.2 |
Fra-2, isoform 2 |
|
 |
T26121 |
|
| |
1.1.2.2 |
Subfamily: ATF-3-like factors |
TGACGTCA |
|
| |
1.1.2.2.1 |
ATF-3 |
|
 |
T08977 |
 |
 |
|
| |
1.1.2.2.1.1 |
ATF-3, isoform 1 |
|
 |
T04850 |
|
| |
1.1.2.2.1.2 |
ATF-3, isoform 2 (ATF-3b) |
|
|
T08976 |
|
| |
1.1.2.2.2 |
JDP-2 |
 |
 |
T19034 |
 |
 |
|
| |
1.1.3 |
Family: Maf-related factors |
TGCTGACTCAGCA |
|
| |
1.1.3.1 |
Subfamily: Large Maf factors |
TGCTGACTCAGCA |
|
| |
1.1.3.1.1 |
c-Maf |
|
 |
T01432 |
 |
 |
|
| |
1.1.3.1.1.1 |
c-Maf, isoform 1 |
|
 |
T09212 |
|
| |
1.1.3.1.1.2 |
c-Maf, isoform 2 |
|
 |
T26181 |
|
| |
1.1.3.1.1.3 |
c-Maf, isoform 3 |
|
 |
T26182 |
|
| |
1.1.3.1.2 |
MafA |
|
 |
T08226 |
 |
 |
|
| |
1.1.3.1.3 |
MafB |
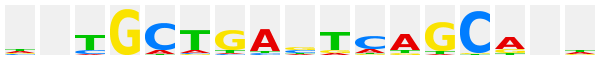 |
 |
T01439 |
 |
 |
 |
| |
1.1.3.1.4 |
Nrl |
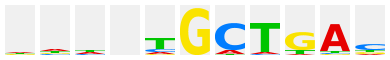 |
 |
T01438 |
 |
 |
|
| |
1.1.3.2 |
Subfamily: Small Maf factors |
TGCTGACTCAGCA |
|
| |
1.1.3.2.1 |
MafF |
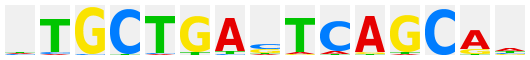 |
 |
T05751 |
 |
 |
|
| |
1.1.3.2.2 |
MafG |
 |
 |
T05750 |
 |
 |
|
| |
1.1.3.2.3 |
MafK |
 |
 |
T01435 |
 |
 |
|
| |
1.1.4 |
Family: B-ATF-related factors |
TGAGTCA |
|
| |
1.1.4.0.1 |
B-ATF |
|
 |
T19854 |
 |
 |
|
| |
1.1.4.0.2 |
B-ATF-2 |
|
 |
T |
 |
 |
|
| |
1.1.4.0.2.1 |
B-ATF-2, isoform 1 |
|
 |
T |
|
| |
1.1.4.0.2.2 |
B-ATF-2, isoform 2 |
|
 |
T |
|
| |
1.1.4.0.3 |
B-ATF-3 |
 |
 |
T18807 |
 |
 |
|
| |
1.1.5 |
Family: XBP-1-related factors |
GGATGACGTGTACA |
|
| |
1.1.5.0.1 |
XBP-1 |
 |
 |
T08152 |
 |
 |
|
| |
1.1.5.0.1.1 |
XBP-1U |
|
 |
T19362 |
|
| |
1.1.5.0.1.2 |
XBP-1S |
|
 |
T11222 |
|
| |
1.1.6 |
Family: ATF-4-related factors |
TGACGTCA |
|
| |
1.1.6.0.1 |
ATF-4 |
 |
 |
T08444 |
 |
 |
|
| |
1.1.6.0.2 |
ATF-5 |
|
 |
T16632 |
 |
 |
|
| |
1.1.7 |
Family: CREB-related factors |
TGACGTCA |
|
| |
1.1.7.1 |
Subfamily: CREB-like factors |
TGACGTCA |
|
| |
1.1.7.1.1 |
CREB (CREB-1) |
|
 |
T02280 |
 |
 |
 |
| |
1.1.7.1.1.1 |
CREB, isoform 1 (CREBα) |
|
 |
T00989 |
|
| |
1.1.7.1.1.2 |
CREB, isoform 2 (CREBΔ) |
|
 |
T01311 |
|
| |
1.1.7.1.1.3 |
CREB, isoform 3 (CREBβ) |
|
|
T02361 |
|
| |
1.1.7.1.1.4 |
CREB, isoform 4 (CREBω) |
|
|
T01312 |
|
| |
1.1.7.1.2 |
ATF-1 |
|
 |
T01304 |
 |
 |
|
| |
1.1.7.1.3 |
CREM |
|
 |
T10306 |
 |
 |
|
| |
1.1.7.1.3.1 |
CREM-τ, isoform 1 (CREM-BCEFGγHIβ) |
|
 |
T01320 |
|
| |
1.1.7.1.3.2 |
CREM-α, isoform 2 |
|
 |
T01314 |
|
| |
1.1.7.1.3.3 |
CREM-β, isoform 3 |
|
 |
T01315 |
|
| |
1.1.7.1.3.4 |
CREM-γ, isoform 4 |
|
 |
T01316 |
|
| |
1.1.7.1.3.5 |
CREM-ταγ, isoform 5 |
|
 |
T26061 |
|
| |
1.1.7.1.3.6 |
CREM-τ2α, isoform 6 |
|
 |
T26062 |
|
| |
1.1.7.1.3.7 |
CREM-τ1α, isoform 7 |
|
 |
T26063 |
|
| |
1.1.7.1.3.8 |
CREM-τ1αγ, isoform 8 |
|
 |
T26064 |
|
| |
1.1.7.1.3.9 |
CREM-τ1γ, isoform 9 |
|
 |
T26065 |
|
| |
1.1.7.1.3.10 |
CREM-αγ, isoform 10 |
|
 |
T26376 |
|
| |
1.1.7.2 |
Subfamily: CREB-3-like factors |
TGACGTCA |
|
| |
1.1.7.2.1 |
CREB-3 (Luman, LZIP) |
 |
 |
T18876 |
 |
 F F |
|
| |
1.1.7.2.1.1 |
CREB-3, isoform 1 (LZIP-1) |
|
 |
T18875 |
|
| |
1.1.7.2.1.2 |
LZIP, isoform 2 (LZIP-2) |
|
 |
T18874 |
|
| |
1.1.7.2.2 |
CREB-3L1 (OASIS) |
 |
 |
T18855 |
 |
 |
|
| |
1.1.7.2.2.1 |
CREB-3L1, isoform 1 |
|
 |
T18854 |
|
| |
1.1.7.2.2.2 |
CREB-3L1, isoform 2 |
|
 |
T18853 |
|
| |
1.1.7.2.3 |
CREB-3L2 |
 |
 |
T15600 |
 |
 |
|
| |
1.1.7.2.4 |
CREB-3L3 (CREB-H) |
|
 |
T18861 |
 |
 |
|
| |
1.1.7.2.5 |
CREB-3L4 (AIBZIP) |
|
 |
T18868 |
 |
 |
|
| |
1.1.7.2.5.1 |
CREB-3L4, isoform 1 (Tisp40β) |
|
 |
T18866 |
|
| |
1.1.7.2.5.2 |
CREB-3L4, isoform 2 (Tisp40α) |
|
 |
T18867 |
|
| |
1.1.7.3 |
Subfamily: ATF-6 factors |
TGACGTGG |
|
| |
1.1.7.3.1 |
ATF-6α |
|
 |
T26785 |
 |
 |
|
| |
1.1.7.3.2 |
ATF-6β |
|
 |
T18795 |
 |
 |
|
| |
1.1.7.4 |
Subfamily: CREBZF-like factors |
TGACGTCA |
|
| |
1.1.7.4.1 |
CREBZF (Zhangfei, ZF) |
|
 |
T |
 |
 |
|
| |
1.1.7.5 |
Subfamily: CREBL2-like factors |
TGACGTCA |
|
| |
1.1.7.5.1 |
CREBL2 |
|
 |
T19897 |
 |
 |
|
| |
1.1.8 |
Family: C/EBP-related |
ATTGCGCAAT |
|
| |
1.1.8.1 |
Subfamily: C/EBP |
ATTGCGCAAT |
|
| |
1.1.8.1.1 |
C/EBPα |
|
 |
T08464 |
 |
 |
 |
| |
1.1.8.1.2 |
C/EBPβ (NF-IL6-1) |
 |
 |
T09409 |
 |
 |
 |
| |
1.1.8.1.2.1 |
C/EBPβ, isoform 1 (C/EBPβ-FL) |
|
 |
T00017 |
|
| |
1.1.8.1.2.2 |
C/EBPβ, isoform 2 (C/EBPβ-LAP) |
|
 |
|
|
| |
1.1.8.1.2.3 |
C/EBPβ, isoform 3 (C/EBPβ-LIP) |
|
 |
T02360 |
|
| |
1.1.8.1.3 |
C/EBPγ |
 |
 |
T00216 |
 |
 |
|
| |
1.1.8.1.4 |
C/EBPδ |
 |
 |
T01114 |
 |
 |
|
| |
1.1.8.1.5 |
C/EBPε |
 |
 |
T16653 |
 |
 |
|
| |
1.1.8.1.6 |
DDIT3 (CHOP-10) |
|
 |
T00127 |
 |
 |
|
| |
1.1.8.2 |
Subfamily: PAR factors |
TTATGCAA |
|
| |
1.1.8.2.1 |
DBP |
 |
 |
T05865 |
 |
 |
|
| |
1.1.8.2.2 |
HLF |
 |
 |
T19989 |
 |
 |
|
| |
1.1.8.2.3 |
TEF |
 |
 |
T10579 |
 |
 |
|
| |
1.1.8.2.3.1 |
TEF-α |
|
 |
T10575 |
|
| |
1.1.8.2.3.2 |
TEF-β |
|
 |
T10576 |
|
| |
1.1.8.2.3.3 |
TEF, isoform 2 |
|
 |
T10577 |
|
| |
1.1.8.2.4 |
NF-IL3 (E4BP4) |
 |
 |
T16464 |
 |
 |
|
| |
1.1.0 |
Family: ZIP only |
|
|
| |
1.1.0.1 |
Subfamily: TSC22 |
|
|
| |
1.1.0.1.1 |
TSC22D1 (TSC-22, Cerebral protein 2) |
|
 |
T27854 |
 |
 |
|
| |
1.1.0.1.1.1 |
TSC22D1, isoform 1 |
|
 |
T27936 |
|
| |
1.1.0.1.1.2 |
TSC22D1, isoform 2 |
|
 |
T27360 |
|
| |
1.1.0.1.1.3 |
TSC22D1, isoform 3 |
|
 |
T27937 |
|
| |
1.1.0.1.2 |
TSC22D2 (TILZ4) |
|
 |
T |
 |
 |
|
| |
1.1.0.1.3 |
TSC22D3 (DSIPI, GILZ) |
|
 |
T22155 |
 |
 |
|
| |
1.1.0.1.3.1 |
TSC22D3, isoform 1 (Tilz3b) |
|
 |
T25737 |
|
| |
1.1.0.1.3.2 |
TSC22D3, isoform 2 (Tilz3a) |
|
 |
T25738 |
|
| |
1.1.0.1.3.3 |
TSC22D3, isoform 3 (Tilz3c) |
|
 |
T25739 |
|
| |
1.1.0.1.4 |
TSC22D4 (TILZ2) |
|
 |
T |
 |
 |
|
| |
1.1.0.2 |
Subfamily: UTF |
|
|
| |
1.1.0.2.1 |
UTF1 |
|
 |
T09523 |
 |
 |
|
| |
1.1.0.2.1.1 |
UTF1, isoform 1 |
|
 |
T08886 |
|
| |
1.1.0.2.1.2 |
UTF1, isoform 2 |
|
 |
T26349 |
|
| |
1.2 |
Class: Basic helix-loop-helix factors (bHLH) |
 |
 |
| |
1.2.1 |
Family: E2A-related factors |
CAGGTG |
|
| |
1.2.1.0.1 |
E2A (TCF-3, ITF-1) |
 |
 |
T10541 |
 |
 |
 |
| |
1.2.1.0.1.1 |
E12 (PAN-2) |
|
 |
T01786 |
|
| |
1.2.1.0.1.2 |
E47 (PAN-1) |
|
 |
T01788 |
|
| |
1.2.1.0.2 |
SEF2 (E2-2, TCF-4, ITF-2, MITF) |
 |
 |
T19028 |
 |
 |
|
| |
1.2.1.0.2.1 |
SEF2, isoform 1 (MITF-2A) |
|
 |
T19025 |
|
| |
1.2.1.0.2.2 |
SEF2, isoform 2 (MITF-2B) |
|
 |
T19027 |
|
| |
1.2.1.0.2.3 |
SEF2, isoform 3 (MITF-2BΔ) |
|
 |
T19026 |
|
| |
1.2.1.0.3 |
HTF-4 (TCF-12, HEB) |
|
 |
T09513 |
 |
 |
|
| |
1.2.1.0.3.1 |
HTF-4a, isoform 1 (ME1A; α) |
|
 |
T01496 |
|
| |
1.2.1.0.3.2 |
HTF-4b, isoform 2 (ME1B; γ) |
|
 |
T01497 |
|
| |
1.2.2 |
Family: MyoD / ASC-related factors |
CAGGTG |
|
| |
1.2.2.1 |
Subfamily: Myogenic transcription factors |
|
|
| |
1.2.2.1.1 |
MyoD (Myod1) |
|
 |
T00506 |
 |
 |
 |
| |
1.2.2.1.2 |
Myogenin (Myog, Myf-4) |
|
 |
T00528 |
 |
 |
|
| |
1.2.2.1.3 |
Myf-5 |
|
 |
T01534 |
 |
 |
|
| |
1.2.2.1.4 |
Myf-6 (MRF4) |
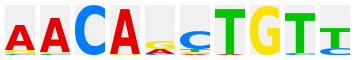 |
 |
T16555 |
 |
 |
|
| |
1.2.2.2 |
Subfamily: Achaete-Scute-like factors |
|
|
| |
1.2.2.2.1 |
ASH-1 (ASCL1) |
|
 |
T01619 |
 |
 |
|
| |
1.2.2.2.2 |
ASH-2 (ASCL2) |
 |
 |
T16775 |
 |
 |
|
| |
1.2.2.2.3 |
ASH-3 (ASCL3) |
|
 |
T19843 |
 |
 |
|
| |
1.2.2.2.4 |
ASH-4 (ASCL4) |
|
 |
T |
 |
 |
|
| |
1.2.2.2.5 |
ASH-5 (ASCL5) |
|
 |
T |
 |
 |
|
| |
1.2.3 |
Family: Tal-related factors |
CAGCTG |
|
| |
1.2.3.1 |
Subfamily: Tal / HEN-like factors |
CAGCTG |
|
| |
1.2.3.1.1 |
Tal-1 (SCL, TCL-5) |
|
 |
T16151 |
 |
 |
|
| |
1.2.3.1.2 |
Tal-2 |
|
 |
T01631 |
 |
 |
|
| |
1.2.3.1.3 |
Lyl-1 |
|
 |
T |
 |
 |
|
| |
1.2.3.1.4 |
HEN1 (NSCL-1, NHLH1) |
 |
 |
T01655 |
 |
 |
|
| |
1.2.3.1.5 |
HEN2 (NSCL-2, NHLH2) |
|
 |
T01657 |
 |
 |
|
| |
1.2.3.2 |
Subfamily: Twist-like factors |
CGTCTG |
|
| |
1.2.3.2.1 |
Twist-1 (H-Twist, M-Twist) |
|
 |
T01635 |
 |
 |
|
| |
1.2.3.2.2 |
Twist-2 (Dermo-1) |
|
 |
T02404 |
 |
 |
|
| |
1.2.3.2.3 |
HAND-1 (eHAND, Th1) |
|
 |
T01569 |
 |
 |
|
| |
1.2.3.2.4 |
HAND-2 (dHAND) |
|
 |
T04370 |
 |
 |
|
| |
1.2.3.2.5 |
bHLH-EC2 (TCF-15) |
|
 |
T01613 |
 |
 |
|
| |
1.2.3.2.6 |
Scx |
|
 |
T19232 |
 |
 |
|
| |
1.2.3.2.7 |
PTF-1a |
|
 |
T01227 |
 |
 |
|
| |
1.2.3.2.8 |
FIGα |
 |
 |
T15614 |
 |
 |
|
| |
1.2.3.3 |
Subfamily: Mesp-like factors |
|
|
| |
1.2.3.3.1 |
Mesp-1 |
 |
 |
T20013 |
 |
 |
|
| |
1.2.3.3.2 |
Mesp-2 |
|
 |
T06095 |
 |
 |
|
| |
1.2.3.3.3 |
TCF-23 |
|
 |
T |
 |
 |
|
| |
1.2.3.3.4 |
ABF-1 (Musculin, MyoR, MSC) |
 |
 |
T05193 |
 |
 |
|
| |
1.2.3.3.5 |
Pod-1 (TCF-21) |
 |
 |
T19300 |
 |
 |
|
| |
1.2.3.3.6 |
Mesogenin-1 (MSGN1) |
|
 |
T20026 |
 |
 |
|
| |
1.2.3.4 |
Subfamily: Neurogenin / Atonal-like factors |
CAGCTG |
|
| |
1.2.3.4.1 |
NeuroD1 |
|
 |
T01626 |
 |
 |
 |
| |
1.2.3.4.2 |
NeuroD2 |
 |
 |
T17539 |
 |
 |
|
| |
1.2.3.4.3 |
NeuroD4 (ATH-3) |
|
 |
T20046 |
 |
 |
|
| |
1.2.3.4.4 |
NeuroD6 (ATH-2) |
|
 |
T01629 |
 |
 |
|
| |
1.2.3.4.5 |
NGN-1 (NeuroD3) |
|
 |
T08093 |
 |
 |
|
| |
1.2.3.4.6 |
NGN-2 (Atoh4, NeuroG2) |
 |
 |
T08094 |
 |
 |
|
| |
1.2.3.4.7 |
NGN-3 (Atoh5, NeuroG3) |
|
 |
T06058 |
 |
 |
|
| |
1.2.3.4.8 |
ATH-1 (ATOH-1) |
 |
 |
T01668 |
 |
 |
|
| |
1.2.3.4.9 |
ATH-5 (ATOH-7) |
|
 |
T18802 |
 |
 |
|
| |
1.2.3.4.10 |
ATH-6 (ATOH-8) |
|
 |
T19852 |
 |
 |
|
| |
1.2.3.4.11 |
bHLHe22 (Beta3) |
 |
 |
T |
 |
 |
|
| |
1.2.3.4.12 |
bHLHe23 |
 |
 |
T |
 |
 |
|
| |
1.2.3.4.13 |
OLIGO1 |
 |
 |
T05853 |
 |
 |
|
| |
1.2.3.4.14 |
OLIGO2 |
 |
 |
T05852 |
 |
 |
|
| |
1.2.3.4.15 |
OLIGO3 |
 |
 |
T20054 |
 |
 |
|
| |
1.2.3.4.16 |
MIST1 (bHLHa15) |
 |
 |
T |
 |
 |
|
| |
1.2.3.4.17 |
Fer3L (Ferd3L) |
|
 |
T19925 |
 |
 |
|
| |
1.2.3.5 |
Subfamily: bHLHa9-like factors |
|
|
| |
1.2.3.5.1 |
bHLHa9 |
|
 |
T |
 |
 |
|
| |
1.2.4 |
Family: Hairy-related factors |
CACGAG |
|
| |
1.2.4.1 |
Subfamily: Hairy-like factors |
CACGAG |
|
| |
1.2.4.1.1 |
HES-1 |
|
 |
T09896 |
 |
 |
|
| |
1.2.4.1.2 |
HES-2 |
|
 |
T |
 |
 |
|
| |
1.2.4.1.3 |
HES-3 |
|
 |
T05081 |
 |
 |
|
| |
1.2.4.1.5 |
HES-5 |
 |
 |
T18965 |
 |
 |
|
| |
1.2.4.1.6 |
HES-6 |
|
 |
T27825 |
 |
 |
|
| |
1.2.4.1.7 |
HES-7 |
 |
 |
T19984 |
 |
 |
|
| |
1.2.4.1.8 |
HEY-1 |
 |
 |
T05077 |
 |
 |
|
| |
1.2.4.1.9 |
HEY-2 |
 |
 |
T05083 |
 |
 |
|
| |
1.2.4.1.10 |
HEYL (HRT3) |
|
 |
T05082 |
 |
 |
|
| |
1.2.4.1.11 |
HELT |
|
 |
T16559 |
 |
 |
|
| |
1.2.4.1.12 |
DEC1 (STRA-13, bHLHb2) |
 |
 |
T02327 |
 |
 |
|
| |
1.2.4.1.13 |
DEC2 (bHLHb3) |
 |
 |
T05845 |
 |
 |
|
| |
1.2.5 |
Family: PAS domain factors |
CACGC |
|
| |
1.2.5.1 |
Subfamily: Ahr-like factors |
CACGC |
|
| |
1.2.5.1.1 |
AhR |
|
 |
T00194 |
 |
 |
|
| |
1.2.5.1.2 |
AhRR |
|
 |
T18746 |
 |
 |
|
| |
1.2.5.1.2.1 |
AhRR, isoform 1 |
|
 |
T05429 |
|
| |
1.2.5.1.2.2 |
AhRR, isoform 2 |
|
 |
T26012 |
|
| |
1.2.5.1.2.3 |
AhRR, isoform 3 |
|
 |
T26013 |
|
| |
1.2.5.1.3 |
SIM1 |
|
 |
T20110 |
 |
 |
|
| |
1.2.5.1.4 |
SIM2 |
|
 |
T19235 |
 |
 |
|
| |
1.2.5.1.5 |
HIF-1α |
|
 |
T04666 |
 |
 |
|
| |
1.2.5.1.5.1 |
HIF-1α, isoform 1 |
|
 |
T18977 |
|
| |
1.2.5.1.5.2 |
HIF-1α, isoform 2 |
|
 |
T26140 |
|
| |
1.2.5.1.6 |
HIF-3α |
|
 |
T18986 |
 |
 |
|
| |
1.2.5.1.6.1 |
HIF-3α, isoform 1 |
|
 |
T18988 |
|
| |
1.2.5.1.6.2 |
HIF-3α, isoform 2 |
|
 |
T18987 |
|
| |
1.2.5.1.7 |
EPAS-1 |
|
 |
T02719 |
 |
 |
|
| |
1.2.5.1.8 |
NPAS-1 |
|
 |
T05876 |
 |
 |
|
| |
1.2.5.1.9 |
NPAS-3 |
|
 |
T05878 |
 |
 F F |
|
| |
1.2.5.2 |
Subfamily: Arnt-like factors |
CACGTG |
|
| |
1.2.5.2.1 |
Arnt (HIF-1β) |
|
 |
T01797 |
 |
 |
|
| |
1.2.5.2.1.1 |
Arnt long, isoform 1 |
|
 |
T10458 |
|
| |
1.2.5.2.1.2 |
Arnt short, isoform 2 |
|
 |
T18790 |
|
| |
1.2.5.2.2 |
Arnt-2 |
|
 |
T18787 |
 |
 |
|
| |
1.2.5.2.2.1 |
Arnt-2, isoform 1 |
|
 |
T18785 |
|
| |
1.2.5.2.2.2 |
Arnt-2, isoform 2 |
|
 |
T18786 |
|
| |
1.2.5.2.3 |
ArntL (Bmal1) |
 |
 |
T05869 |
 |
 |
|
| |
1.2.5.2.3.1 |
ArntL, isoform 1 (b') |
|
 |
T10228 |
|
| |
1.2.5.2.3.2 |
ArntL, isoform 2 (b) |
|
 |
T18818 |
|
| |
1.2.5.2.3.3 |
ArntL, isoform 3 |
|
 |
T18819 |
|
| |
1.2.5.2.3.4 |
ArntL, isoform 4 |
|
 |
T18820 |
|
| |
1.2.5.2.3.5 |
ArntL, isoform 5 (g') |
|
 |
T18821 |
|
| |
1.2.5.2.4 |
ArntL2 (Bmal2) |
|
 |
T05874 |
 |
 |
|
| |
1.2.5.2.4.1 |
ArntL2, isoform 1 (Bmal2a) |
|
 |
T11140 |
|
| |
1.2.5.2.4.2 |
ArntL2, isoform 2 (Bmal2b) |
|
 |
T26045 |
|
| |
1.2.5.2.5 |
CLOCK |
 |
 |
T05868 |
 |
 |
|
| |
1.2.5.2.5.1 |
CLOCK, isoform Long |
|
 |
T10259 |
|
| |
1.2.5.2.5.2 |
CLOCK, isoform Short |
|
 |
T18843 |
|
| |
1.2.5.2.6 |
NPAS-2 |
|
 |
T05877 |
 |
 |
|
| |
1.2.5.3 |
Subfamily: NCoA factors |
|
|
| |
1.2.5.3.1 |
NCoA-1 (SRC-1) |
|
 |
T04639 |
 |
 |
|
| |
1.2.5.3.1.1 |
NCoA-1, isoform 1 (SRC-1A) |
|
 |
T25725 |
|
| |
1.2.5.3.1.2 |
NCoA-1, isoform 2 (SRC-1E) |
|
 |
T09181 |
|
| |
1.2.5.3.1.3 |
NCoA-1, isoform 3 |
|
 |
T26206 |
|
| |
1.2.5.3.1.4 |
NCoA-1, isoform 4 |
|
 |
T26207 |
|
| |
1.2.5.3.2 |
NCoA-2 |
|
 |
T02482 |
 |
 |
|
| |
1.2.5.3.3 |
NCoA-3 |
|
 |
T04638 |
 |
 |
|
| |
1.2.5.4 |
Subfamily: NPAS-4 |
CACGA |
|
| |
1.2.5.4.1 |
NPAS-4 |
|
 |
T |
 |
 |
|
| |
1.2.5.5 |
Subfamily: SOHLH-like factors |
|
|
| |
1.2.5.5.1 |
SOHLH1 (TEB2) |
|
 |
T20117 |
 |
 |
|
| |
1.2.5.5.2 |
SOHLH2 (TEB1) |
|
 |
T19246 |
 |
 |
|
| |
1.2.5.5.3 |
TCFL5 (Cha, E2BP-1) |
|
 |
T17742 |
 |
 F F |
|
| |
1.2.6 |
Family: bHLH-ZIP factors |
CACATG |
|
| |
1.2.6.1 |
Subfamily: TFE3-like factors |
CACATG |
|
| |
1.2.6.1.1 |
TFE3 (TFEA) |
 |
 |
T09192 |
 |
 |
|
| |
1.2.6.1.1.1 |
TFE3, isoform 1 |
|
 |
T19333 |
|
| |
1.2.6.1.1.2 |
TFE3, isoform 2 |
|
 |
T26336 |
|
| |
1.2.6.1.1.3 |
TFE3, isoform 3 |
|
 |
T26337 |
|
| |
1.2.6.1.1.4 |
TFE3, isoform 4 |
|
 |
T26338 |
|
| |
1.2.6.1.2 |
TFEB |
 |
 |
T06082 |
 |
 |
|
| |
1.2.6.1.3 |
TFEC |
 |
 |
T14666 |
 |
 |
|
| |
1.2.6.1.4 |
MiTF |
|
 |
T01554 |
 |
 |
|
| |
1.2.6.1.4.1 |
MiTF, isoform A |
|
 |
T19067 |
|
| |
1.2.6.1.4.2 |
MiTF, isoform A1 |
|
 |
T19068 |
|
| |
1.2.6.1.4.3 |
MiTF, isoform A2 |
|
 |
T19066 |
|
| |
1.2.6.1.4.4 |
MiTF, isoform H |
|
 |
T19065 |
|
| |
1.2.6.1.4.5 |
MiTF, isoform H1 |
|
 |
T19069 |
|
| |
1.2.6.1.4.6 |
MiTF, isoform H2 |
|
 |
T19070 |
|
| |
1.2.6.1.4.7 |
MiTF, isoform H3 |
|
 |
T19071 |
|
| |
1.2.6.1.4.8 |
MiTF, isoform M |
|
 |
T09287 |
|
| |
1.2.6.1.4.9 |
MiTF, isoform M1 |
|
 |
T19072 |
|
| |
1.2.6.2 |
Subfamily: USF factors |
CACGTG |
|
| |
1.2.6.2.1 |
USF-1 |
 |
 |
T00877 |
 |
 |
 |
| |
1.2.6.2.2 |
USF-2 |
|
 |
T01555 |
 |
 |
|
| |
1.2.6.2.2.1 |
USF-2A |
|
 |
T08315 |
|
| |
1.2.6.2.2.2 |
USF-2B |
|
 |
T11000 |
|
| |
1.2.6.3 |
Subfamily: SREBP factors |
ATCACCCCAC |
|
| |
1.2.6.3.1 |
SREBP-1 (SREBF1) |
 |
 |
T10488 |
 |
 |
 |
| |
1.2.6.3.1.1 |
SREBP-1A |
|
 |
T15263 |
|
| |
1.2.6.3.1.2 |
SREBP-1A-W42 |
|
 |
T26305 |
|
| |
1.2.6.3.1.3 |
SREBP-1C |
|
 |
T08890 |
|
| |
1.2.6.3.1.4 |
SREBP-1C-W42 |
|
 |
T26306 |
|
| |
1.2.6.3.2 |
SREBP-2 (SREBF2) |
 |
 |
T15631 |
 |
 |
|
| |
1.2.6.3.2.1 |
SREBP-2, isoform 1 |
|
 |
T19272 |
|
| |
1.2.6.3.2.2 |
SREBP-2, isoform 2 |
|
 |
T26307 |
|
| |
1.2.6.4 |
Subfamily: AP-4 |
CAGCTG |
|
| |
1.2.6.4.1 |
AP-4 |
 |
 |
T16526 |
 |
 |
|
| |
1.2.6.5 |
Subfamily: Myc / Max factors |
CACGTG |
|
| |
1.2.6.5.1 |
c-Myc |
|
 |
T00143 |
 |
 |
 |
| |
1.2.6.5.2 |
N-Myc |
|
 |
T01445 |
 |
 |
|
| |
1.2.6.5.3 |
L-Myc-1 |
|
 |
T02387 |
 |
 |
|
| |
1.2.6.5.5 |
Max |
 |
 |
T16608 |
 |
 |
 |
| |
1.2.6.5.6 |
B-Myc |
|
 |
T10695 |
|
 |
|
| |
1.2.6.6 |
Subfamily: Mondo-like factors |
CACGTG |
|
| |
1.2.6.6.1 |
Mlx (Tcfl4) |
 |
 |
T05123 |
 |
 |
|
| |
1.2.6.6.1.1 |
Mlxα |
|
 |
T19092 |
|
| |
1.2.6.6.1.2 |
Mlxβ |
|
 |
T08952 |
|
| |
1.2.6.6.1.3 |
Mlxγ |
|
 |
T19093 |
|
| |
1.2.6.6.2 |
MondoA (Mlx-IP) |
|
 |
T05125 |
 |
 |
|
| |
1.2.6.6.2.1 |
Mlx-IP, isoform 1 |
|
 |
T18651 |
|
| |
1.2.6.6.2.2 |
Mlx-IP, isoform 2 |
|
 |
T18652 |
|
| |
1.2.6.6.3 |
MondoB (Mlx-IPL, WS-bHLH) |
 |
 |
T05122 |
 |
 |
|
| |
1.2.6.6.3.1 |
Mlx-IPL, isoform 1 (ζ) |
|
 |
T14593 |
|
| |
1.2.6.6.3.2 |
Mlx-IPL, isoform 2 (ϑ) |
|
 |
T26195 |
|
| |
1.2.6.6.3.3 |
Mlx-IPL, isoform 3 (ι) |
|
 |
T26196 |
|
| |
1.2.6.6.3.4 |
Mlx-IPL, isoform 4 (κ) |
|
 |
T26197 |
|
| |
1.2.6.6.3.5 |
Mlx-IPL, isoform 5 (η) |
|
 |
T26198 |
|
| |
1.2.6.7 |
Subfamily: Mad-like factors |
CACGTG |
|
| |
1.2.6.7.1 |
Mad1 (MXD1) |
|
 |
T02390 |
 |
 |
 |
| |
1.2.6.7.2 |
Mad3 (MXD3) |
|
 |
T02391 |
 |
 |
|
| |
1.2.6.7.3 |
Mad4 (MXD4) |
|
 |
T02392 |
 |
 |
|
| |
1.2.6.7.4 |
Mad5 (MGA) = 6.5.5.0.2 |
|
 |
T03940 |
 |
 |
|
| |
1.2.6.7.4.1 |
Mad5, isoform 1 |
|
 |
T14744 |
|
| |
1.2.6.7.4.2 |
Mad5, isoform 2 |
|
 |
T26191 |
|
| |
1.2.6.7.4.3 |
Mad5, isoform 3 |
|
 |
T26192 |
|
| |
1.2.6.7.4.4 |
Mad5, isoform 4 |
|
 |
T26193 |
|
| |
1.2.6.7.5 |
Mxi-1 |
|
 |
T09074 |
 |
 |
|
| |
1.2.6.7.5.1 |
Mxi-1, isoform 1 (long) |
|
 |
T09075 |
|
| |
1.2.6.7.5.2 |
Mxi-1, isoform 2 (short) |
|
 |
T09076 |
|
| |
1.2.6.7.6 |
Mnt |
 |
 |
T03269 |
 |
 F F |
|
| |
1.2.8 |
Family: HLH domain only |
|
|
| |
1.2.8.0.1 |
Id1 |
|
 |
T00403 |
 |
 |
|
| |
1.2.8.0.1.1 |
Id1, isoform Long |
|
 |
T25726 |
|
| |
1.2.8.0.1.2 |
Id1, isoform Short |
|
 |
T09185 |
|
| |
1.2.8.0.2 |
Id2 |
|
 |
T00404 |
 |
 |
|
| |
1.2.8.0.3 |
Id3 |
|
 |
T00367 |
 |
 |
|
| |
1.2.8.0.4 |
Id4 |
 |
 |
T01658 |
 |
 |
|
| |
1.3 |
Class: Basic helix-span-helix factors (bHSH) |
 |
 |
| |
1.3.1 |
Family: AP-2 |
GCCTGAGGC |
|
| |
1.3.1.0.1 |
AP-2α |
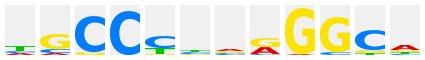 |
 |
T10025 |
 |
 |
|
| |
1.3.1.0.1.1 |
AP-2α, isoform 1 |
|
 |
T00033 |
|
| |
1.3.1.0.1.2 |
AP-2α, isoform 2 |
|
 |
T02488 |
|
| |
1.3.1.0.1.3 |
AP-2α, isoform 3 |
|
 |
T02489 |
|
| |
1.3.1.0.1.4 |
AP-2α, isoform 4 |
|
 |
T02490 |
|
| |
1.3.1.0.2 |
AP-2β |
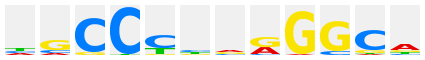 |
 |
T02469 |
 |
 |
|
| |
1.3.1.0.3 |
AP-2γ |
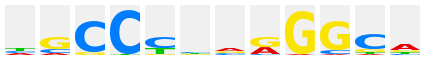 |
 |
T02470 |
 |
 |
|
| |
1.3.1.0.4 |
AP-2δ |
|
 |
T06431 |
 |
 |
|
| |
1.3.1.0.5 |
AP-2ε |
|
 |
T16752 |
 |
 |
|
|
| 2 |
Superclass: Zinc-coordinating DNA-binding domains |
 |
| |
2.1 |
Class: Nuclear receptors with C4 zinc fingers |
 |
 |
| |
2.1.1 |
Family: Steroid hormone receptors (NR3) |
|
|
| |
2.1.1.1 |
Subfamily: GR-like receptors (NR3C) |
AGAACATGATGTTCT |
|
| |
2.1.1.1.1 |
Glucocorticoid receptor (GR) (NR3C1) |
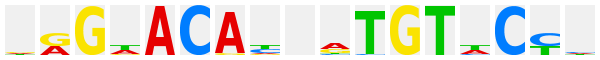 |
 |
T00335 |
 |
 |
 |
| |
2.1.1.1.1.1 |
GR, isoform 1 (A, 1-A) |
|
 |
T08555 |
|
| |
2.1.1.1.1.2 |
GR, isoform 2 (B, 2-A) |
|
 |
T18951 |
|
| |
2.1.1.1.1.3 |
GR, isoform 1-B |
|
 |
|
|
| |
2.1.1.1.1.4 |
GR, isoform 2-B |
|
 |
|
|
| |
2.1.1.1.2 |
Mineralocorticoid receptor (MR) (NR3C2) |
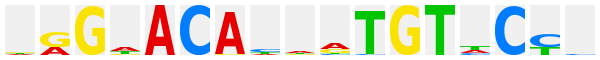 |
 |
T17766 |
 |
 |
|
| |
2.1.1.1.3 |
Progesterone receptor (PR) (NR3C3) |
|
 |
T04680 |
 |
 |
 |
| |
2.1.1.1.4 |
Androgen receptor (AR) (NR3C4) |
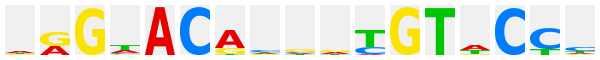 |
 |
T00041 |
 |
 |
 |
| |
2.1.1.2 |
Subfamily: ER-like receptors (NR3A&B) |
AGGTCACAGTGACCT |
|
| |
2.1.1.2.1 |
Estrogen receptor α (ERα) (ESR1, NR3A1) |
 |
 |
T00259 |
 |
 |
 |
| |
2.1.1.2.2 |
Estrogen receptor β (ERβ) (NR3A2) |
|
 |
T17427 |
 |
 |
|
| |
2.1.1.2.2.1 |
ERβ1, isoform 1 |
|
 |
T18909 |
|
| |
2.1.1.2.2.2 |
ERβ2, isoform 2 |
|
 |
T18910 |
|
| |
2.1.1.2.2.3 |
ERβ2A, isoform 3 (5A) |
|
 |
T18912 |
|
| |
2.1.1.2.2.4 |
ERβ3, isoform 4 |
|
 |
T26417 |
|
| |
2.1.1.2.2.5 |
ERβ4, isoform 5 |
|
 |
T26418 |
|
| |
2.1.1.2.3 |
ERR1 (ERRα, ESRRA) (NR3B1) |
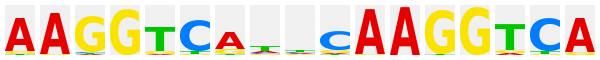 |
 |
T04849 |
 |
 |
|
| |
2.1.1.2.4 |
ERR2 (ERRβ, ESRRB) (NR3B2) |
 |
 |
T06175 |
 |
 |
 |
| |
2.1.1.2.5 |
ERR3 (ERRγ, ESRRG) (NR3B3) |
 |
 |
T14585 |
 |
 |
|
| |
2.1.1.2.5.1 |
ERR3, isoform 1 |
|
 |
T04644 |
|
| |
2.1.1.2.5.2 |
ERR3, isoform 2 |
|
 |
T04647 |
|
| |
2.1.2 |
Family: Thyroid hormone receptor-related factors (NR1) |
|
|
| |
2.1.2.1 |
Subfamily: Retinoic acid receptors (NR1B) |
TGACCTTTGACCT |
|
| |
2.1.2.1.1 |
RAR-α (NR1B1) |
 |
 |
T01327 |
 |
 |
 |
| |
2.1.2.1.1.1 |
RAR-α1 |
|
 |
T01335 |
|
| |
2.1.2.1.1.2 |
RAR-α2 |
|
 |
T01336 |
|
| |
2.1.2.1.2 |
RAR-β (NR1B2) |
 |
 |
T01328 |
 |
 |
|
| |
2.1.2.1.2.1 |
RAR-β1 |
|
 |
T01337 |
|
| |
2.1.2.1.2.2 |
RAR-β2 |
|
 |
T01338 |
|
| |
2.1.2.1.2.3 |
RAR-β3 |
|
 |
T01339 |
|
| |
2.1.2.1.2.4 |
RAR-β4 |
|
 |
T01361 |
|
| |
2.1.2.1.3 |
RAR-γ (NR1B3) |
 |
 |
T01329 |
 |
 |
|
| |
2.1.2.1.3.1 |
RAR-γ, isoform 1 (A) |
|
 |
T01340 |
|
| |
2.1.2.1.3.2 |
RAR-γ, isoform 2 (B) |
|
 |
T26259 |
|
| |
2.1.2.1.3.3 |
RAR-γ, isoform 3 |
|
 |
T26260 |
|
| |
2.1.2.2 |
Subfamily: Thyroid hormone receptors (NR1A) |
TGACCTGAAATGACCT |
|
| |
2.1.2.2.1 |
T3R-α (THRA) (NR1A1) |
 |
 |
T08894 |
 |
 |
|
| |
2.1.2.2.1.1 |
T3R-α1 |
|
 |
T00836 |
|
| |
2.1.2.2.1.2 |
T3R-α2 |
|
 |
T01173 |
|
| |
2.1.2.2.1.3 |
T3R-α3 |
|
 |
T17546 |
|
| |
2.1.2.2.1.4 |
T3R-αΔE6 |
|
 |
T26340 |
|
| |
2.1.2.2.2 |
T3R-β (NR1A2) |
 |
 |
T08896 |
 |
 |
 |
| |
2.1.2.2.2.1 |
T3R-β1 |
|
 |
T04722 |
|
| |
2.1.2.2.2.2 |
T3R-β2 |
|
 |
T04723 |
|
| |
2.1.2.3 |
Subfamily: Rev-ErbA (NR1D) |
TGACCTAGTGACCT |
|
| |
2.1.2.3.1 |
Rev-ErbAα (NR1D1) |
|
 |
T06117 |
 |
 |
|
| |
2.1.2.3.2 |
Rev-ErbAβ (NR1D2) |
|
 |
T04896 |
 |
 |
|
| |
2.1.2.4 |
Subfamily: Vitamin D receptor (NR1I) |
TGACCTTCGTGACCT |
|
| |
2.1.2.4.1 |
VDR (NR1I1) |
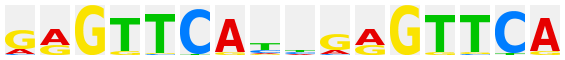 |
 |
T00883 |
 |
 |
 |
| |
2.1.2.4.2 |
PXR (NR1I2) |
|
 |
T08235 |
 |
 |
|
| |
2.1.2.4.2.1 |
PXR, isoform 1 (PXR.1) |
|
 |
T04618 |
|
| |
2.1.2.4.2.2 |
PXR, isoform 2 (PXR.2) |
|
 |
T04619 |
|
| |
2.1.2.4.3 |
CAR (NR1I3) |
|
 |
T05687 |
 |
 |
|
| |
2.1.2.4.3.1 |
CAR1 |
|
 |
T08261 |
|
| |
2.1.2.4.3.2 |
CAR2 |
|
 |
T02755 |
|
| |
2.1.2.5 |
Subfamily: PPAR (NR1C) |
TGACCTTTGACCT |
|
| |
2.1.2.5.1 |
PPARα (NR1C1) |
|
 |
T00694 |
 |
 |
|
| |
2.1.2.5.2 |
PPARβ (PPARδ) (NR1C2) |
|
 |
T04781 |
 |
 |
 |
| |
2.1.2.5.3 |
PPARγ (NR1C3) |
|
 |
T05347 |
 |
 |
 |
| |
2.1.2.5.3.1 |
PPARγ1, isoform 1 |
|
 |
T02529 |
|
| |
2.1.2.5.3.2 |
PPARγ2, isoform 2 |
|
 |
T05332 |
|
| |
2.1.2.6 |
Subfamily: ROR (NR1F) |
TGACCTACTTAT |
|
| |
2.1.2.6.1 |
RORα (NR1F1) |
 |
 |
T06119 |
 |
 F F |
|
| |
2.1.2.6.1.1 |
RORα1 |
|
 |
T09284 |
|
| |
2.1.2.6.1.2 |
RORα2 |
|
 |
|
|
| |
2.1.2.6.1.3 |
RORα3 |
|
 |
|
|
| |
2.1.2.6.1.4 |
RORα4 |
|
 |
T15650 |
|
| |
2.1.2.6.2 |
RORβ (NR1F2) |
|
 |
T17581 |
 |
 |
|
| |
2.1.2.6.2.1 |
RORβ, isoform 1 |
|
 |
T19213 |
|
| |
2.1.2.6.2.2 |
RORβ, isoform 2 |
|
 |
T17683 |
|
| |
2.1.2.6.3 |
RORγ (NR1F3) |
|
 |
T10351 |
 |
 |
|
| |
2.1.2.6.3.1 |
RORγ, isoform 1 |
|
 |
T06118 |
|
| |
2.1.2.6.3.2 |
RORγ, isoform 2 (RORgT) |
|
 |
T10350 |
|
| |
2.1.2.7 |
Subfamily: LXR (NR1H) |
TGACCTCTACTGACCT |
|
| |
2.1.2.7.1 |
LXRα (NR1H3) |
|
 |
T04430 |
 |
 |
|
| |
2.1.2.7.2 |
LXRβ (NR1H2) |
|
 |
T04467 |
 |
 |
|
| |
2.1.2.7.3 |
FXR (NR1H4) |
|
 |
T02753 |
 |
 |
|
| |
2.1.2.7.3.1 |
FXR, isoform 1 |
|
 |
T09890 |
|
| |
2.1.2.7.3.2 |
FXR, isoform 2 |
|
 |
T09299 |
|
| |
2.1.3 |
Family: RXR-related receptors (NR2) |
(TGACCT) |
|
| |
2.1.3.1 |
Subfamily: Retinoid X receptors (NR2B) |
(TGACCT) |
|
| |
2.1.3.1.1 |
RXRα (NR2B1) |
 |
 |
T01331 |
 |
 |
 |
| |
2.1.3.1.2 |
RXRβ (NR2B2) |
 |
 |
T01332 |
 |
 F F |
|
| |
2.1.3.1.2.1 |
RXRβ1, isoform long |
|
 |
T02779 |
|
| |
2.1.3.1.2.2 |
RXRβ2, isoform short |
|
 |
T01366 |
|
| |
2.1.3.1.3 |
RXRγ (NR2B3) |
 |
 |
T01333 |
 |
 |
|
| |
2.1.3.2 |
Subfamily: HNF-4 (NR2A) |
AGTCCAAAGTTCA |
|
| |
2.1.3.2.1 |
HNF-4α (NR2A1) |
 |
 |
T01181 |
 |
 |
 |
| |
2.1.3.2.1.1 |
HNF-4α, isoform long |
|
 |
T28928 |
|
| |
2.1.3.2.1.2 |
HNF-4α, isoform short |
|
 |
T26145 |
|
| |
2.1.3.2.2 |
HNF-4γ (NR2A2) |
|
 |
T06235 |
 |
 |
|
| |
2.1.3.3 |
Subfamily: Tailless-like receptors (NR2E) |
TGACCTTTGACCT |
|
| |
2.1.3.3.1 |
Tlx (NR2E1) |
 |
 |
T02357 |
 |
 |
|
| |
2.1.3.3.2 |
PNR (NR2E3) |
|
 |
T04861 |
 |
|
|
| |
2.1.3.4 |
Subfamily: Testicular receptors (NR2C) |
TGACCTCTGACCT |
|
| |
2.1.3.4.1 |
TR2 (NR2C1) |
|
 |
T04847 |
 |
 |
|
| |
2.1.3.4.1.1 |
TR2-11, isoform 1 |
|
 |
T11074 |
|
| |
2.1.3.4.1.2 |
TR2-9, isoform 2 (TR2-11-t) |
|
 |
T11075 |
|
| |
2.1.3.4.2 |
TR4 (NR2C2) |
 |
 |
T16549 |
 |
 |
|
| |
2.1.3.5 |
Subfamily: COUP-like receptors (NR2F) |
TGACCTTTGACCT |
|
| |
2.1.3.5.1 |
COUP-TFI (NR2F1) |
 |
 |
T00994 |
 |
 |
 |
| |
2.1.3.5.2 |
COUP-TFII (NR2F2) |
|
 |
T06154 |
 |
 |
|
| |
2.1.3.5.3 |
EAR2 (NR2F6) |
 |
 |
T06163 |
 |
 |
|
| |
2.1.4 |
Family: NGFI-B-related receptors (NR4) |
TGACCTTT |
|
| |
2.1.4.0.1 |
NGFI-B (Nur77, NR4A1) |
|
 |
T01348 |
 |
 |
 |
| |
2.1.4.0.2 |
NURR1 (NR4A2) |
 |
 |
T10226 |
 |
 |
|
| |
2.1.4.0.2.1 |
NURR1, isoform 1 |
|
 |
T04312 |
|
| |
2.1.4.0.2.2 |
NURR1, isoform 2 (NURR1A) |
|
 |
T10247 |
|
| |
2.1.4.0.3 |
NOR1 (NR4A3, TEC) |
|
 |
T15963 |
 |
 |
|
| |
2.1.4.0.3.1 |
NOR1, isoform α (long) |
|
 |
T19145 |
|
| |
2.1.4.0.3.2 |
NOR1, isoform β (short, TECΔC) |
|
 |
T19146 |
|
| |
2.1.5 |
Family: FTZ-F1-related receptors (NR5) |
TGACCTTG |
|
| |
2.1.5.0.1 |
FTZ-F1 (SF-1) (NR5A1) |
|
 |
T08551 |
 |
 |
 |
| |
2.1.5.0.1.1 |
FTZ-F1, isoform 1 |
|
 |
T09519 |
|
| |
2.1.5.0.1.2 |
FTZ-F1, isoform 2 |
|
 |
T25132 |
|
| |
2.1.5.0.2 |
LRH-1 (NR5A2) |
|
 |
T04754 |
 |
 |
|
| |
2.1.6 |
Family: GCNF-related receptors (NR6) |
TGAACTTGACTTGA |
|
| |
2.1.6.0.1 |
GCNF (NR6A1) |
|
 |
T15701 |
 |
 F F |
|
| |
2.1.6.0.1.1 |
GCNF, isoform 1 |
|
 |
T02772 |
|
| |
2.1.6.0.1.2 |
GCNF, isoform 2 |
|
 |
T26225 |
|
| |
2.1.6.0.1.3 |
GCNF, isoform 3 |
|
 |
T26226 |
|
| |
2.1.7 |
Family: DAX-related receptors (NR0) |
TGACCTT |
|
| |
2.1.7.0.1 |
DAX1 (NR0B1) |
|
 |
T04677 |
 |
 |
|
| |
2.1.7.0.2 |
SHP (NR0B2) |
|
 |
T05321 |
 |
 |
|
| |
2.2 |
Class: Other C4 zinc finger-type factors |
 |
 |
| |
2.2.1 |
Family: GATA-type zinc fingers |
|
|
| |
2.2.1.1 |
Subfamily: Two zinc-finger GATA factors |
AGATAA |
|
| |
2.2.1.1.1 |
GATA-1 (GF-1, EryF1, NF-E1) |
|
 |
T00305 |
 |
 |
 |
| |
2.2.1.1.1.1 |
GATA-1, isoform 1 |
|
 |
T08291 |
|
| |
2.2.1.1.1.2 |
GATA-1, isoform 2 (GATA-1s) |
|
 |
T26130 |
|
| |
2.2.1.1.2 |
GATA-2 |
|
 |
T01302 |
 |
 |
|
| |
2.2.1.1.3 |
GATA-3 |
 |
 |
T00310 |
 |
 |
|
| |
2.2.1.1.4 |
GATA-4 |
 |
 |
T02532 |
 |
 |
|
| |
2.2.1.1.5 |
GATA-5 |
 |
 |
T02685 |
 |
 |
|
| |
2.2.1.1.6 |
GATA-6 |
|
 |
T02686 |
 |
 |
|
| |
2.2.1.1.6.1 |
GATA-6, isoform 1 |
|
 |
T10208 |
|
| |
2.2.1.1.6.2 |
GATA-6, isoform 2 |
|
 |
T26132 |
|
| |
2.2.1.2 |
Subfamily: Single GATA-type zinc-finger |
AGATAA |
|
| |
2.2.1.2.1 |
GATAD1 (ODAG) |
|
 |
T |
 |
 |
|
| |
2.2.1.2.1.1 |
GATAD1, isoform 1 |
|
 |
T |
|
| |
2.2.1.2.1.2 |
GATAD1, isoform 2 |
|
 |
T |
|
| |
2.2.1.2.2 |
p66-α (GATAD2A) |
|
 |
T |
 |
 |
|
| |
2.2.1.2.3 |
p66-β (GATAD2B) |
|
 |
T |
 |
 |
|
| |
2.2.1.2.3.1 |
p66-β, isoform 1 |
|
 |
T |
|
| |
2.2.1.2.3.2 |
p66-β, isoform 2 |
|
 |
T |
|
| |
2.2.1.2.4 |
RERE (ARG, ARP, ATN1L) = 3.5.1.3.7 |
|
 |
T |
 |
 |
|
| |
2.2.1.2.5 |
MTA1 = 3.5.1.3.4 |
|
 |
T27821 |
 |
 |
|
| |
2.2.1.2.6 |
MTA2 (MTA1L1, PID) = 3.5.1.3.5 |
|
 |
T |
 |
 |
|
| |
2.2.1.2.7 |
MTA3 = 3.5.1.3.6 |
|
 |
T27832 |
 |
 F F |
|
| |
2.2.1.2.7.1 |
MTA3, isoform 1 |
|
 |
T27227 |
|
| |
2.2.1.2.7.2 |
MTA3, isoform 2 |
|
 |
T27228 |
|
| |
2.2.1.2.8 |
ZGLP1 (GLP-1) |
|
 |
T |
 |
 |
|
| |
2.2.1.2.9 |
DNMT3A |
|
 |
T27827 |
 |
 |
|
| |
2.2.1.2.9.1 |
DNMT3A, isoform 1 |
|
 |
T27196 |
|
| |
2.2.1.2.9.2 |
DNMT3A, isoform 2 |
|
 |
T27868 |
|
| |
2.2.1.2.10 |
DNMT3B |
|
 |
T |
 |
 |
|
| |
2.2.1.2.10.1 |
DNMT3B, isoform 1 |
|
 |
T |
|
| |
2.2.1.2.10.2 |
DNMT3B, isoform 2 |
|
 |
T |
|
| |
2.2.1.2.10.3 |
DNMT3B, isoform 3 |
|
 |
T |
|
| |
2.2.1.2.10.4 |
DNMT3B, isoform 4 |
|
 |
T |
|
| |
2.2.1.2.11 |
DNMT3L |
|
 |
T |
 |
 |
|
| |
2.2.1.2.12 |
ATRX (RAD54L, XH2) |
|
 |
T |
 |
 |
 |
| |
2.2.1.2.13 |
TRPS1 (GC79) = 2.3.4.0.20 |
|
 |
T19349 |
 |
 |
|
| |
2.3 |
Class: C2H2 zinc finger factors |
 |
 |
| |
2.3.1 |
Family: Three-zinc finger Krüppel-related factors |
|
|
| |
2.3.1.1 |
Subfamily: Sp1-like factors |
GGGGCGGGG |
|
| |
2.3.1.1.1 |
Sp1 |
 |
 |
T00752 |
 |
 |
 |
| |
2.3.1.1.1.1 |
Sp1, isoform 1 |
|
 |
T09118 |
|
| |
2.3.1.1.1.2 |
Sp1, isoform 2 (Sp1-S) |
|
 |
T11472 |
|
| |
2.3.1.1.2 |
Sp2 |
|
 |
T06543 |
 |
 |
|
| |
2.3.1.1.2.1 |
Sp2, isoform 1 |
|
 |
T10134 |
|
| |
2.3.1.1.2.2 |
Sp2, isoform 2 |
|
 |
T10135 |
|
| |
2.3.1.1.3 |
Sp3 |
 |
 |
T05840 |
 |
 F F |
|
| |
2.3.1.1.3.1 |
Sp3, isoform 1 (L-Sp3) |
|
 |
T19262 |
|
| |
2.3.1.1.3.2 |
Sp3, isoform 2 |
|
 |
T19261 |
|
| |
2.3.1.1.3.3 |
Sp3, isoform 3 (M1-Sp3) |
|
 |
T26303 |
|
| |
2.3.1.1.4 |
Sp4 |
 |
 |
T02414 |
 |
 |
|
| |
2.3.1.1.5 |
Sp5 |
|
 |
T19265 |
 |
 |
|
| |
2.3.1.1.6 |
Sp6 |
|
 |
T05074 |
 |
 |
|
| |
2.3.1.1.7 |
Sp7 |
|
 |
T20132 |
 |
 |
|
| |
2.3.1.1.8 |
Sp8 |
 |
 |
T27853 |
 |
 |
|
| |
2.3.1.1.9 |
Sp9 |
|
 |
T |
 |
 |
|
| |
2.3.1.1.9.1 |
Sp9, isoform 1 |
|
 |
T |
|
| |
2.3.1.1.9.2 |
Sp9, isoform 2 |
|
 |
T |
|
| |
2.3.1.2 |
Subfamily: Krüppel-like factors |
CCACACCCT |
|
| |
2.3.1.2.1 |
KLF1 (EKLF) |
|
 |
T01676 |
 |
 |
|
| |
2.3.1.2.2 |
KLF2 (LKLF) |
|
 |
T01677 |
 |
 |
|
| |
2.3.1.2.3 |
KLF3 (BKLF) |
|
 |
T10980 |
 |
 |
 |
| |
2.3.1.2.4 |
KLF4 (GKLF) |
|
 |
T02450 |
 |
 |
 |
| |
2.3.1.2.5 |
KLF5 (CKLF, IKLF, BTEB2) |
|
 |
T05054 |
 |
 |
 |
| |
2.3.1.2.6 |
KLF6 (CPBP, BCD1) |
|
 |
T19036 |
 |
 |
|
| |
2.3.1.2.7 |
KLF7 (UKLF) |
|
 |
T26756 |
 |
 F F |
|
| |
2.3.1.2.8 |
KLF8 (BKLF) |
|
 |
T20006 |
 |
 |
|
| |
2.3.1.2.9 |
KLF9 (BTEB1) |
|
 |
T04700 |
 |
 |
|
| |
2.3.1.2.10 |
KLF10 (TIEG1) |
|
 |
T15844 |
 |
 |
 |
| |
2.3.1.2.11 |
KLF11 (FKLF, TIEG2) |
|
 |
T17849 |
 |
 |
|
| |
2.3.1.2.12 |
KLF12 (AP-2rep) |
 |
 |
T04701 |
 |
 |
|
| |
2.3.1.2.13 |
KLF13 (BTEB3, NSLP1) |
 |
 |
T05052 |
 |
 |
|
| |
2.3.1.2.14 |
KLF14 (BTEB5) |
 |
 |
T20000 |
 |
 |
|
| |
2.3.1.2.15 |
KLF15 (KKLF) |
|
 |
T20002 |
 |
 |
 |
| |
2.3.1.2.16 |
KLF16 (BTEB4, NSLP2) |
 |
 |
T05053 |
 |
 |
|
| |
2.3.1.2.17 |
KLF17 |
|
 |
T20004 |
 |
 |
|
| |
2.3.1.3 |
Subfamily: EGR factors |
GCGTGGGCG |
|
| |
2.3.1.3.1 |
EGR1 (Krox-24, Zif268) |
 |
 |
T10013 |
 |
 |
 |
| |
2.3.1.3.2 |
EGR2 (Krox-20) |
 |
 |
T00454 |
 |
 |
|
| |
2.3.1.3.2.1 |
EGR2, isoform long |
|
 |
T25981 |
|
| |
2.3.1.3.2.2 |
EGR2, isoform short |
|
 |
T08340 |
|
| |
2.3.1.3.3 |
EGR3 (PILOT) |
 |
 |
T05539 |
 |
 |
|
| |
2.3.1.3.4 |
EGR4 |
 |
 |
T19919 |
 |
 |
|
| |
2.3.2 |
Family: Other factors with up to three adjacent zinc fingers |
|
|
| |
2.3.2.1 |
Subfamily: Factors with 2-3 adjacent zinc fingers
and a BTB/POZ domain |
TCCTGCTA |
|
| |
2.3.2.1.1 |
ZBTB5 [2] |
|
 |
T19397 |
 |
 |
|
| |
2.3.2.1.2 |
ZBTB8B [2] |
|
 |
T |
 |
 |
|
| |
2.3.2.1.6 |
ZBTB22 (ZNF297, BING1) [3] |
|
 |
T19377 |
 |
 |
|
| |
2.3.2.1.7 |
ZBTB32 (ZNF538, FAZF) [3] |
|
 |
T15957 |
 |
 |
|
| |
2.3.2.1.7.1 |
ZBTB32, isoform 1 |
|
 |
T15956 |
|
| |
2.3.2.1.7.2 |
ZBTB32, isoform 2 |
|
 |
T15958 |
|
| |
2.3.2.1.8 |
ZBTB34 [3] |
|
 |
T |
 |
 |
|
| |
2.3.2.1.9 |
ZBTB37 [3] |
|
 |
T |
 |
 |
|
| |
2.3.2.1.10 |
ZBTB43 (ZBTB22B, ZNF297B) [3] |
|
 |
T19385 |
 |
 |
 |
| |
2.3.2.1.11 |
ZBTB46 (BTBD4, ZNF340) [2] |
|
 |
T |
 |
 |
|
| |
2.3.2.1.11.1 |
ZBTB46, isoform 1 |
|
 |
T |
|
| |
2.3.2.1.11.2 |
ZBTB46, isoform 2 |
|
 |
T |
|
| |
2.3.2.1.12 |
ZBTB33 (KAISO, ZNF348) [3] |
|
 |
T08299 |
 |
 |
|
| |
2.3.2.2 |
Subfamily: Factors with 2-3 adjacent zinc fingers
and a KRAB domain |
|
|
| |
2.3.2.3 |
Subfamily: Factors with 2-3 adjacent zinc fingers
and a SCAN domain |
|
|
| |
2.3.2.4 |
Subfamily: Other three adjacent zinc finger factors |
|
|
| |
2.3.2.4.1 |
ZNF414 (Zfp414) [3] |
|
 |
T20584 |
 |
 |
|
| |
2.3.2.4.1.1 |
ZNF414, isoform 1 |
|
 |
T20583 |
|
| |
2.3.2.4.1.2 |
ZNF414, isoform 2 |
|
 |
T20585 |
|
| |
2.3.2.4.2 |
ZNF511 (Zfp511) [3] |
|
 |
T |
 |
 |
|
| |
2.3.2.4.2.1 |
ZNF511, isoform 1 |
|
 |
T |
|
| |
2.3.2.4.2.2 |
ZNF511, isoform 2 |
|
 |
T |
|
| |
2.3.2.4.3 |
ZNF580 (Zfp580) [3] |
|
 |
T19527 |
 |
 |
|
| |
2.3.2.4.5 |
ZNF740 (ZFP740) [3] |
 |
 |
T16895 |
 |
 |
|
| |
2.3.2.4.5.1 |
ZNF740, isoform 1 |
|
 |
T16894 |
|
| |
2.3.2.4.5.2 |
ZNF740, isoform 2 |
|
 |
T16897 |
|
| |
2.3.2.4.5.3 |
ZNF740, isoform 3 |
|
 |
T16898 |
|
| |
2.3.2.4.6 |
ZFPM2 (ZNF89B, FOG2) = 2.7.2.0.2 [3] |
|
 |
T18922 |
 |
 F F |
|
| |
2.3.2.4.6.1 |
ZFPM2, isoform 1 |
|
 |
T18923 |
|
| |
2.3.2.4.6.2 |
ZFPM2, isoform 2 |
|
 |
T18924 |
|
| |
2.3.2.4.6.3 |
ZFPM2, isoform 3 |
|
 |
T18925 |
|
| |
2.3.2.4.7 |
OSR1 (ODD) [3] |
|
 |
T16932 |
 |
 |
|
| |
2.3.2.4.8 |
OVOL3 (OVL2L) [4] |
|
 |
T |
 |
 F, ? F, ? |
|
| |
2.3.2.4.9 |
AEBP2 [2] |
|
 |
T18741 |
 |
 |
|
| |
2.3.2.4.9.1 |
AEBP2, isoform 1 |
|
 |
T18740 |
|
| |
2.3.2.4.9.2 |
AEBP2, isoform 2 |
|
 |
T18736 |
|
| |
2.3.2.4.9.3 |
AEBP2, isoform 3 |
|
 |
T18737 |
|
| |
2.3.2.4.9.4 |
AEBP2, isoform 4 |
|
 |
T18738 |
|
| |
2.3.2.4.9.5 |
AEBP2, isoform 5 |
|
 |
T18739 |
|
| |
2.3.3 |
Family: More than 3 adjacent zinc finger factors |
|
|
| |
2.3.3.1 |
Subfamily: GLI-like factors |
TGGGTGGTC |
|
| |
2.3.3.1.1 |
GLI1 [5] |
|
 |
T06385 |
 |
 |
 |
| |
2.3.3.1.2 |
GLI2 [5] |
 |
 |
T06386 |
 |
 |
|
| |
2.3.3.1.3 |
GLI3 [5] |
|
 |
T06387 |
 |
 |
|
| |
2.3.3.1.4 |
GLIS1 [5] |
 |
 |
T06390 |
 |
 |
|
| |
2.3.3.1.4.1 |
GLIS1, isoform 1 |
|
 |
T11184 |
|
| |
2.3.3.1.4.2 |
GLIS1, isoform 2 |
|
 |
T26134 |
|
| |
2.3.3.1.5 |
GLIS2 (NKL) [5] |
 |
 |
T |
 |
 |
|
| |
2.3.3.1.6 |
GLIS3 (ZNF515) [5] |
 |
 |
T17410 |
 |
 |
|
| |
2.3.3.1.6.1 |
GLIS3, isoform 1 |
|
 |
T18954 |
|
| |
2.3.3.1.6.2 |
GLIS3, isoform 2 |
|
 |
T15909 |
|
| |
2.3.3.1.7 |
ZIC1 (ZNF201) [5] |
 |
 |
T04669 |
 |
 |
|
| |
2.3.3.1.8 |
ZIC2 [5] |
|
 |
T04670 |
 |
 |
|
| |
2.3.3.1.9 |
ZIC3 (ZNF203) [5] |
 |
 |
T04671 |
 |
 |
 |
| |
2.3.3.1.9.1 |
ZIC3, isoform 1 (ZIC3A) |
|
 |
T14517 |
|
| |
2.3.3.1.9.2 |
ZIC3, isoform 2 (ZIC3B) |
|
 |
T26531 |
|
| |
2.3.3.1.10 |
ZIC4 [5] |
 |
 |
T |
 |
 F F |
|
| |
2.3.3.1.11 |
ZIC5 [4] |
|
 |
T |
 |
 |
|
| |
2.3.3.2 |
Subfamily: Snail-like factors |
CACCTG |
|
| |
2.3.3.2.1 |
SNAI1 [4] |
|
 |
T02333 |
 |
 |
|
| |
2.3.3.2.2 |
SNAI2 (SLUG) [5] |
 |
 |
T19238 |
 |
 |
|
| |
2.3.3.2.3 |
SNAI3 (ZNF293) [5] |
|
 |
T10589 |
 |
 |
|
| |
2.3.3.2.4 |
SCRT1 [5] |
 |
 |
T20104 |
 |
 |
|
| |
2.3.3.2.5 |
SCRT2 [5] |
 |
 |
T20108 |
 |
 |
|
| |
2.3.3.3 |
Subfamily: ZFP91-like factors |
|
|
| |
2.3.3.3.1 |
ZFP91 (ZNF757) [5] |
|
 |
T19408 |
 |
 |
|
| |
2.3.3.3.1.1 |
ZFP91, isoform 1 |
|
 |
T19407 |
|
| |
2.3.3.3.1.2 |
ZFP91, isoform 2 |
|
 |
T26362 |
|
| |
2.3.3.3.2 |
ZNF692 [5] |
|
 |
T19554 |
 |
 |
 |
| |
2.3.3.3.3 |
ZNF276 (ZFP276, ZNF477) [5] |
|
 |
T20488 |
 |
 |
|
| |
2.3.3.3.3.1 |
ZNF276, isoform 1 |
|
 |
T20487 |
|
| |
2.3.3.3.3.2 |
ZNF276, isoform 2 |
|
 |
T20489 |
|
| |
2.3.3.3.4 |
ZNF653 (ZIP67) [5] |
|
 |
T20934 |
 |
 |
|
| |
2.3.3.3.4.1 |
ZNF653, isoform 1 |
|
 |
T20933 |
|
| |
2.3.3.3.4.2 |
ZNF653, isoform 2 |
|
 |
T20935 |
|
| |
2.3.3.4 |
Subfamily: ZNF280-like zinc finger factors |
|
|
| |
2.3.3.4.2 |
ZNF280B (SUHW2, ZNF279, ZNF632, Zfp280b) [4] |
|
 |
T |
 |
 |
|
| |
2.3.3.4.3 |
ZNF280C (SUHW3, ZNF633, Zfp280c) [5] |
|
 |
T20167 |
 |
 |
|
| |
2.3.3.4.4 |
ZNF280D (SUHW4, ZNF634) [5] |
|
 |
T20177 |
 |
 |
|
| |
2.3.3.4.4.1 |
ZNF280D, isoform 1 |
|
 |
T20176 |
|
| |
2.3.3.4.4.2 |
ZNF280D, isoform 2 |
|
 |
T20178 |
|
| |
2.3.3.4.4.3 |
ZNF280D, isoform 3 |
|
 |
T20179 |
|
| |
2.3.3.4.5 |
POGZ (SUHW5, ZNF280E, ZNF635) [9] |
|
 |
T |
 |
 |
 |
| |
2.3.3.5 |
Subfamily: ZSCAN5-like zinc finger factors |
|
|
| |
2.3.3.6 |
Subfamily: ZNF75-like zinc finger factors |
|
|
| |
2.3.3.7 |
Subfamily: ZNF705 factors |
|
|
| |
2.3.3.8 |
Subfamily: ZBTB7 factors |
GACCCC |
|
| |
2.3.3.8.1 |
ZBTB7A [4] |
 |
 |
T08304 |
 |
 |
|
| |
2.3.3.8.1.1 |
ZBTB7A, isoform 1 |
|
 |
T19395 |
|
| |
2.3.3.8.1.2 |
ZBTB7A, isoform 2 |
|
 |
T08303 |
|
| |
2.3.3.8.2 |
ZBTB7B [4] |
 |
 |
T09541 |
 |
 |
|
| |
2.3.3.8.3 |
ZBTB7C [4] |
 |
 |
T |
 |
 |
|
| |
2.3.3.9 |
Subfamily: YY1-like factors |
GCCATCTTG |
|
| |
2.3.3.9.1 |
YY1 [4] |
 |
 |
T00278 |
 |
 |
 |
| |
2.3.3.9.2 |
YY2 (ZNF631) [4] |
 |
 |
T17589 |
 |
 |
|
| |
2.3.3.9.3 |
ZFP42 (REX1, ZNF754) [4] |
|
 |
T00727 |
 |
|
|
| |
2.3.3.10 |
Subfamily: ZNF24-like factors |
TCAT |
|
| |
2.3.3.10.1 |
ZNF24 (KOX17, ZNF191, ZSCAN3) [4] |
|
 |
T21161 |
 |
 |
 |
| |
2.3.3.10.6 |
ZKSCAN1 (KOX18, ZNF139, ZNF36) [6] |
|
 |
T20343 |
 |
 |
|
| |
2.3.3.10.6.1 |
ZKSCAN1, isoform 1 |
|
 |
T20342 |
|
| |
2.3.3.10.6.2 |
ZKSCAN1, isoform 2 |
|
 |
T20344 |
|
| |
2.3.3.11 |
Subfamily: ZBTB6-like factors |
|
|
| |
2.3.3.11.1 |
ZBTB6 (ZID, ZNF482) [4] |
|
 |
T19399 |
 |
 |
|
| |
2.3.3.11.2 |
ZBTB26 (ZNF481) [4] |
|
 |
T |
 |
 |
|
| |
2.3.3.11.3 |
ZBTB12 (NG35) [4] |
|
 |
T16838 |
 |
 |
|
| |
2.3.3.12 |
Subfamily: PRDM1-like factors |
AAGTGAAAGT |
|
| |
2.3.3.12.1 |
PRDM1 (BLIMP1) [4] |
 |
 |
T02316 |
 |
 |
|
| |
2.3.3.12.1.1 |
PRDM1, isoform 1 |
|
 |
T14505 |
|
| |
2.3.3.12.1.2 |
PRDM1, isoform 2 (1A) |
|
 |
T26255 |
|
| |
2.3.3.12.1.3 |
PRDM1, isoform 3 (1B) |
|
 |
T26256 |
|
| |
2.3.3.12.1.4 |
PRDM1, isoform 4 (1C) |
|
 |
T26257 |
|
| |
2.3.3.12.1.5 |
PRDM1, isoform 5 (delta exon 7) |
|
 |
T26258 |
|
| |
2.3.3.13 |
Subfamily: ZNF148-like factors |
CACCC |
|
| |
2.3.3.13.1 |
ZNF148 (ZBP89, Zfp148) [4] |
|
 |
T08694 |
 |
 |
|
| |
2.3.3.13.2 |
ZNF281 (GZP1, ZBP99, Zfp281) [4] |
|
 |
T16856 |
 |
|
|
| |
2.3.3.14 |
Subfamily: ZBTB20-like factors |
|
|
| |
2.3.3.14.1 |
ZBTB20 (DPZF, ZNF288, HOF) [5] |
|
 |
T15597 |
 |
 |
|
| |
2.3.3.14.1.1 |
ZBTB20, isoform 1 (HOF-L) |
|
 |
T15595 |
|
| |
2.3.3.14.1.2 |
ZBTB20, isoform 2 (HOF-S) |
|
 |
T15596 |
|
| |
2.3.3.14.2 |
ZBTB45 (ZNF499) [4] |
|
 |
T |
 |
 |
|
| |
2.3.3.15 |
Subfamily: ZNF524-like factors |
|
|
| |
2.3.3.15.1 |
ZNF524 (Zfp524) [4] |
 |
 |
T20724 |
 |
|
|
| |
2.3.3.16 |
Subfamily: ZNF238-like factors |
AACATCTGGA |
|
| |
2.3.3.16.1 |
ZNF238 (RP58, TAZ1, ZBTB18) [4] |
 |
 |
T16607 |
 |
 |
|
| |
2.3.3.16.2 |
ZBTB42 [4] |
|
 |
T |
 |
 |
|
| |
2.3.3.16.2.1 |
ZBTB42, isoform 1 |
|
 |
T |
|
| |
2.3.3.16.2.2 |
ZBTB42, isoform 2 |
|
 |
T |
|
| |
2.3.3.17 |
Subfamily: OVOL-factors |
GCGGGGG |
|
| |
2.3.3.17.1 |
OVOL1 [4] |
|
 |
T08547 |
 |
 |
|
| |
2.3.3.17.2 |
OVOL2 (mOVO, ZNF339) [4] |
|
 |
T08543 |
 |
 |
|
| |
2.3.3.17.2.1 |
OVOL2, isoform 1 (MOVO-A) |
|
 |
T08542 |
|
| |
2.3.3.17.2.2 |
OVOL2, isoform 2 (MOVO-C) |
|
 |
T08545 |
|
| |
2.3.3.17.2.3 |
OVOL2, isoform 3 (MOVO-B) |
|
 |
T08544 |
|
| |
2.3.3.18 |
Subfamily: ZNF56-like factors |
|
|
| |
2.3.3.19 |
Subfamily: ZNF500-like factors |
|
|
| |
2.3.3.19.3 |
ZKSCAN2 (ZNF694) [6] |
|
 |
T |
 |
 |
|
| |
2.3.3.19.4 |
ZSCAN29 (ZNF690) [6] |
|
 |
T |
 |
|
|
| |
2.3.3.20 |
Subfamily: FEZF factors |
|
|
| |
2.3.3.20.1 |
FEZF1 (ZNF312B) [6] |
|
 |
T19931 |
 |
 |
|
| |
2.3.3.20.2 |
FEZF2 (FEZL, ZNF312) [6] |
|
 |
T19936 |
 |
 |
|
| |
2.3.3.21 |
Subfamily: GFI1 factors |
AAATCACAGC |
|
| |
2.3.3.21.1 |
GFI1 (ZNF163) [6] |
|
 |
T06593 |
 |
 |
 |
| |
2.3.3.21.2 |
GFI1B [6] |
|
 |
T06585 |
 |
 |
|
| |
2.3.3.22 |
Subfamily: BCL6 factors |
CTTTCTAGGAAT |
|
| |
2.3.3.22.1 |
BCL6B (BAZF, ZNF62) [5] |
 |
 |
T16794 |
 |
 |
|
| |
2.3.3.22.2 |
BCL6 (LAZ3, ZBTB27, ZNF51) [6] |
|
 |
T08156 |
 |
 |
|
| |
2.3.3.23 |
Subfamily: ZNF212-like factors |
|
|
| |
2.3.3.24 |
Subfamily: MTF1-like factors |
TGCACAC |
|
| |
2.3.3.24.1 |
MTF1 [6] |
 |
 |
T00515 |
 |
 |
|
| |
2.3.3.24.2 |
ZNF410 (APA1, Zfp410) [5] |
 |
 |
T16880 |
 |
|
|
| |
2.3.3.24.2.1 |
ZNF410, isoform 1 |
|
 |
T16879 |
|
| |
2.3.3.24.2.2 |
ZNF410, isoform 2 |
|
 |
T16883 |
|
| |
2.3.3.24.2.3 |
ZNF410, isoform 3 |
|
 |
T16884 |
|
| |
2.3.3.25 |
Subfamily: PLAG factors |
|
|
| |
2.3.3.25.1 |
PLAGL2 [6] |
|
 |
T18384 |
 |
 |
|
| |
2.3.3.25.2 |
PLAG1 [7] |
|
 |
T21518 |
 |
 |
|
| |
2.3.3.25.3 |
PLAGL1 (LOT1, ZAC) [7] |
|
 |
T10142 |
 |
 |
|
| |
2.3.3.26 |
Subfamily: ZKSCAN3-like factors |
TGAGGGG |
|
| |
2.3.3.26.1 |
ZKSCAN3 (ZFP47, ZNF306, ZNF309, ZSCAN13) [7] |
 |
 |
T |
 |
 |
|
| |
2.3.3.26.2 |
ZKSCAN4 (ZNF307, ZNF427) [7] |
|
 |
T |
 |
 F F |
|
| |
2.3.3.27 |
Subfamily: ZNF525-like factors |
|
|
| |
2.3.3.28 |
Subfamily: ZNF76-like factors |
CCCATCATGCCTTGC |
|
| |
2.3.3.28.1 |
ZNF76 (ZNF523, Zfp523) [7] |
|
 |
T19573 |
 |
 |
|
| |
2.3.3.28.1.1 |
ZNF76, isoform 1 |
|
 |
T25999 |
|
| |
2.3.3.28.1.2 |
ZNF76, isoform 2 |
|
 |
T26000 |
|
| |
2.3.3.28.2 |
ZNF143 (SBF, STAF) [7] |
 |
 |
T18502 |
 |
 |
|
| |
2.3.3.28.2.1 |
ZNF143, isoform 1 |
|
 |
T18501 |
|
| |
2.3.3.28.2.2 |
ZNF143, isoform 2 |
|
 |
T26365 |
|
| |
2.3.3.28.2.3 |
ZNF143, isoform 3 |
|
 |
T26366 |
|
| |
2.3.3.29 |
Subfamily: ZNF652-like factors |
|
|
| |
2.3.3.29.1 |
ZNF652 (ZFP652) [9] |
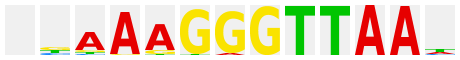 |
 |
T20929 |
 |
 |
|
| |
2.3.3.29.1.1 |
ZNF652, isoform 1 |
|
 |
T20928 |
|
| |
2.3.3.29.1.2 |
ZNF652, isoform 2 |
|
 |
T20930 |
|
| |
2.3.3.30 |
Subfamily: ZNF350-like factors |
TGGgttCAGactTTG |
|
| |
2.3.3.31 |
Subfamily: ZNF642-like factors |
|
|
| |
2.3.3.31.4 |
ZNF583 [12] |
|
 |
T20839 |
 |
|
|
| |
2.3.3.32 |
Subfamily: ZNF679-like factors |
|
|
| |
2.3.3.33 |
Subfamily: ZNF763-like factors |
|
|
| |
2.3.3.34 |
Subfamily: ZNF3-like factors |
|
|
| |
2.3.3.35 |
Subfamily: ZNF620-like factors |
|
|
| |
2.3.3.36 |
Subfamily: ZNF324 factors |
|
|
| |
2.3.3.37 |
Subfamily: ZNF362-like factors |
|
|
| |
2.3.3.38 |
Subfamily: ZNF282-like factors |
TCCACCCC |
|
| |
2.3.3.39 |
Subfamily: ZNF764-like factors |
|
|
| |
2.3.3.39.3 |
ZNF771 [8] |
|
 |
T21051 |
 |
|
|
| |
2.3.3.40 |
Subfamily: ZNF177-like factors |
|
|
| |
2.3.3.41 |
Subfamily: ZNF32-like factors |
|
|
| |
2.3.3.41.1 |
ZNF32 (KOX30, Zfp637) [7] |
|
 |
T |
 |
 |
 |
| |
2.3.3.41.1.1 |
ZNF32, isoform 1 |
|
 |
T |
|
| |
2.3.3.41.1.2 |
ZNF32, isoform 2 |
|
 |
T |
|
| |
2.3.3.41.2 |
ZIK1 (ZNF762) [9] |
|
 |
T20335 |
 |
|
|
| |
2.3.3.42 |
Subfamily: ZNF773-like factors |
|
|
| |
2.3.3.43 |
Subfamily: ZNF558-like factors |
|
|
| |
2.3.3.44 |
Subfamily: ZNF302-like factors |
|
|
| |
2.3.3.44.2 |
ZNF2 (ZNF661, Zfp661) [9] |
|
 |
T21173 |
 |
|
|
| |
2.3.3.45 |
Subfamily: ZNF562-like factors |
|
|
| |
2.3.3.46 |
Subfamily: ZXD-factors |
|
|
| |
2.3.3.46.2 |
ZXDB [10] |
|
 |
T |
 |
|
|
| |
2.3.3.46.3 |
ZXDC (ZXDL) [10] |
|
 |
T |
 |
 |
|
| |
2.3.3.46.3.1 |
ZXDC, isoform 1 |
|
 |
T |
|
| |
2.3.3.46.3.2 |
ZXDC, isoform 2 |
|
 |
T |
|
| |
2.3.3.46.3.3 |
ZXDC, isoform 3 |
|
 |
T |
|
| |
2.3.3.46.3.4 |
ZXDC, isoform 4 |
|
 |
T |
|
| |
2.3.3.47 |
Subfamily: ZNF222-like factors |
|
|
| |
2.3.3.48 |
Subfamily: ZNF736-like factors |
|
|
| |
2.3.3.49 |
Subfamily: ZNF286 factors |
|
|
| |
2.3.3.50 |
Subfamily: CTCF-like factors |
|
|
| |
2.3.3.50.1 |
CTCF [11] |
 |
 |
T02285 |
 |
 |
|
| |
2.3.3.50.2 |
CTCFL (CTCF-T, BORIS) [11] |
|
 |
T |
 |
 |
|
| |
2.3.3.50.2.1 |
CTCFL, isoform 1 |
|
 |
T |
|
| |
2.3.3.50.2.2 |
CTCFL, isoform 2 |
|
 |
T |
|
| |
2.3.3.51 |
Subfamily: ZNF366-like factors |
|
|
| |
2.3.3.51.2 |
ZNF710 (Zfp710) [11] |
|
 |
T21014 |
 |
|
|
| |
2.3.3.51.2.1 |
ZNF710, isoform 1 |
|
 |
T21013 |
|
| |
2.3.3.51.2.2 |
ZNF710, isoform 2 |
|
 |
T21015 |
|
| |
2.3.3.51.2.3 |
ZNF710, isoform 3 |
|
 |
T21016 |
|
| |
2.3.3.52 |
Subfamily: ZNF322-like factors |
|
|
| |
2.3.3.52.1 |
ZNF322A (ZNF322, ZNF388, ZNF489, Zfp322A) [11] |
|
 |
T20185 |
 |
 |
|
| |
2.3.3.53 |
Subfamily: ZNF548-like factors |
|
|
| |
2.3.3.54 |
Subfamily: ZNF460-like factors |
|
|
| |
2.3.3.55 |
Subfamily: ZNF146-like factors |
|
|
| |
2.3.3.55.1 |
ZNF146 [10] |
|
 |
T |
 |
|
|
| |
2.3.3.55.2 |
ZNF260 (ZFP260) [13] |
|
 |
T20476 |
 |
 |
|
| |
2.3.3.56 |
Subfamily: ZNF214-like factors |
|
|
| |
2.3.3.57 |
Subfamily: ZNF479-like factors |
|
|
| |
2.3.3.58 |
Subfamily: ZNF180-like factors |
|
|
| |
2.3.3.58.3 |
ZNF436 (Zfp46) [12] |
|
 |
T20615 |
 |
 |
|
| |
2.3.3.58.6 |
ZSCAN2 (ZFP29, ZNF854) [14] |
|
 |
T21249 |
 |
 |
|
| |
2.3.3.59 |
Subfamily: ZNF100-like factors |
|
|
| |
2.3.3.59.3 |
ZNF431 (Zfp431, Zfp923) [13] |
|
 |
T23370 |
 |
|
|
| |
2.3.3.60 |
Subfamily: ZNF98-like factors |
|
|
| |
2.3.3.61 |
Subfamily: ZNF343-like factors |
|
|
| |
2.3.3.62 |
Subfamily: ZFP2-like factors |
|
|
| |
2.3.3.62.1 |
ZFP2 (ZNF751, Mkr2) [13] |
|
 |
T |
 |
 |
|
| |
2.3.3.62.1.1 |
ZFP2, isoform 1 |
|
 |
|
|
| |
2.3.3.62.1.2 |
ZFP2, isoform 2 |
|
 |
T02335 |
|
| |
2.3.3.63 |
Subfamily: ZFP30-like factors |
|
|
| |
2.3.3.63.1 |
ZFP30 (ZNF745) [13] |
|
 |
T20292 |
 |
|
|
| |
2.3.3.63.2 |
ZFP82 (ZNF545) [13] |
|
 |
T20313 |
 |
 |
|
| |
2.3.3.63.2.1 |
ZFP82, isoform 1 |
|
 |
T20312 |
|
| |
2.3.3.63.2.2 |
ZFP82, isoform 2 |
|
 |
T20314 |
|
| |
2.3.3.63.2.3 |
ZFP82, isoform 3 |
|
 |
T28055 |
|
| |
2.3.3.63.3 |
ZFP14 (ZNF531) [13] |
|
 |
T |
 |
 |
|
| |
2.3.3.64 |
Subfamily: ZNF354A-like factors |
|
|
| |
2.3.3.64.1 |
ZNF354A (EZNF, HKL1, TCF17, Zfp354a) [13] |
|
 |
T19363 |
 |
 |
|
| |
2.3.3.64.2 |
ZNF354B (Zfp354b) [13] |
|
 |
T20196 |
 |
 |
|
| |
2.3.3.65 |
Subfamily: ZFX/ZFY factors |
AGGCCC |
|
| |
2.3.3.65.1 |
ZFX [13] |
|
 |
T16099 |
 |
 |
|
| |
2.3.3.65.2 |
ZFY-1 [13] |
|
 |
T20325 |
 |
|
 |
| |
2.3.3.65.3 |
ZFY-2 [13] |
|
 |
T20327 |
 |
|
 |
| |
2.3.3.66 |
Subfamily: ZNF689-like factors |
|
|
| |
2.3.3.66.1 |
ZNF689 (Zfp689) [12] |
|
 |
T20996 |
 |
 |
|
| |
2.3.3.66.2 |
ZNF672 (Zfp672) [14] |
|
 |
T20968 |
 |
 |
|
| |
2.3.3.67 |
Subfamily: ZFN283-like factors |
|
|
| |
2.3.3.68 |
Subfamily: ZFN81-like factors |
|
|
| |
2.3.3.69 |
Subfamily: ZFN25-like factors |
|
|
| |
2.3.3.69.3 |
ZFP37 [12] |
|
 |
T21364 |
 |
 |
|
| |
2.3.3.70 |
Subfamily: ZFN26-like factors |
|
|
| |
2.3.3.71 |
Subfamily: ZFN300-like factors |
|
|
| |
2.3.3.72 |
Subfamily: ZFN813-like factors |
|
|
| |
2.3.3.73 |
Subfamily: ZNF816A-like factors |
|
|
| |
2.3.3.74 |
Subfamily: ZNF442-like factors |
|
|
| |
2.3.3.75 |
Subfamily: ZNF432-like factors |
|
|
| |
2.3.3.76 |
Subfamily: ZNF665-like factors |
|
|
| |
2.3.3.77 |
Subfamily: ZNF595-like factors |
|
|
| |
2.3.3.78 |
Subfamily: ZNF14-like factors |
|
|
| |
2.3.3.79 |
Subfamily: ZNF841-like factors |
|
|
| |
2.3.3.80 |
Subfamily: ZNF99-like factors |
|
|
| |
2.3.3.0 |
Subfamily: unclassified |
|
|
| |
2.3.3.0.1 |
ZFP161 (ZBTB14, ZNF478, Zfp5) |
CTACCTGCACAGTTCACGGA |
 |
T02349 |
 |
 |
|
| |
2.3.3.0.3 |
ZNF496 (ZKSCAN17, Zfp496) |
|
 |
T |
 |
 |
|
| |
2.3.3.0.3.1 |
ZNF496, isoform 1 |
|
 |
T |
|
| |
2.3.3.0.3.2 |
ZNF496, isoform 2 |
|
 |
T |
|
| |
2.3.3.0.3.3 |
ZNF496, isoform 3 |
|
 |
T |
|
| |
2.3.3.0.9 |
OSR2 |
|
 |
T16872 |
 |
 |
|
| |
2.3.3.0.9.1 |
OSR2, isoform 1 (OSR2A) |
|
 |
T16871 |
|
| |
2.3.3.0.9.2 |
OSR2, isoform 2 (OSR2B) |
|
 |
T16873 |
|
| |
2.3.3.0.13 |
ZNF22 (KOX15, KROX26, Zfp422) |
CAATG |
 |
T21157 |
 |
 |
|
| |
2.3.3.0.17 |
ZFP41 |
|
 |
T20302 |
 |
 |
|
| |
2.3.3.0.20 |
ZNF18 (KOX11, ZKSCAN6, ZNF535, Zfp18, Zfp535) |
|
 |
T21150 |
 |
 |
|
| |
2.3.3.0.21 |
WT1 |
CGCCCCCGC |
 |
T02351 |
 |
 |
 |
| |
2.3.3.0.21.1 |
WT1, isoform 1 |
|
 |
T09249 |
|
| |
2.3.3.0.21.2 |
WT1, isoform 2 |
|
 |
T19358 |
|
| |
2.3.3.0.21.3 |
WT1, isoform 3 |
|
 |
T19356 |
|
| |
2.3.3.0.21.4 |
WT1, isoform 4 |
|
 |
T19357 |
|
| |
2.3.3.0.21.5 |
WT1, isoform 5 |
|
 |
T26353 |
|
| |
2.3.3.0.21.6 |
WT1, isoform 6 |
|
 |
T26354 |
|
| |
2.3.3.0.23 |
PRDM6 (PFM3) |
|
 |
T27850 |
 |
 |
|
| |
2.3.3.0.23.1 |
PRDM6, isoform 1 |
|
 |
T27355 |
|
| |
2.3.3.0.23.2 |
PRDM6, isoform 2 |
|
 |
T27928 |
|
| |
2.3.3.0.23.3 |
PRDM6, isoform 3 |
|
 |
T27929 |
|
| |
2.3.3.0.23.4 |
PRDM6, isoform 4 |
|
 |
T27930 |
|
| |
2.3.3.0.24 |
ZNF444 (EZF2, ZSCAN17, Zfp44) |
|
 |
T |
 |
 |
|
| |
2.3.3.0.25 |
ZNF641 (Zfp641) |
|
 |
T20912 |
 |
 |
|
| |
2.3.3.0.25.1 |
ZNF641, isoform 1 |
|
 |
T20911 |
|
| |
2.3.3.0.25.2 |
ZNF641, isoform 2 |
|
 |
T20913 |
|
| |
2.3.3.0.26 |
ZNF746 (PARIS, Zfp746) |
TATTTTT |
 |
T |
 |
 |
|
| |
2.3.3.0.28 |
ZBTB44 (BTBD15, ZNF851) |
|
 |
T19390 |
 |
 |
|
| |
2.3.3.0.28.1 |
ZBTB44, isoform 1 |
|
 |
T19389 |
|
| |
2.3.3.0.28.2 |
ZBTB44, isoform 2 |
|
 |
T26360 |
|
| |
2.3.3.0.31 |
ZNF655 (VIK, Zfp655) [6] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.34 |
ZNF131 [6] |
|
 |
T23316 |
 |
 |
|
| |
2.3.3.0.34.1 |
ZNF131, isoform 1 |
|
 |
T23324 |
|
| |
2.3.3.0.34.2 |
ZNF131, isoform 2 |
|
 |
T25958 |
|
| |
2.3.3.0.34.3 |
ZNF131, isoform 3 |
|
 |
T26363 |
|
| |
2.3.3.0.35 |
PRDM14 [6] |
GGTCTCTAA |
 |
T |
 |
|
|
| |
2.3.3.0.37 |
ZNF575 (Zfp575) [6] |
|
 |
T20823 |
 |
 |
|
| |
2.3.3.0.38 |
ZBTB49 [7] |
 |
 |
T |
 |
 |
|
| |
2.3.3.0.38.1 |
ZBTB49, isoform 1 |
|
 |
T |
|
| |
2.3.3.0.38.2 |
ZBTB49, isoform 2 |
|
 |
T |
|
| |
2.3.3.0.38.3 |
ZBTB49, isoform 3 |
|
 |
T |
|
| |
2.3.3.0.39 |
ZNF691 (Zfp691) [7] |
|
 |
T16890 |
 |
 |
|
| |
2.3.3.0.40 |
ZNF394 (ZKSCAN14, Zfp394, Zfp99) [7] |
|
 |
T20568 |
 |
 |
|
| |
2.3.3.0.41 |
ZNF449 (ZSCAN19, Zfp449) [7] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.42 |
ZNF498 (ZSCAN25, Zfp498) [7] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.43 |
ZSCAN30 (ZNF397OS, Zfp397OS) [7] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.47 |
ZSCAN21 (ZFP38, ZNF38, Zipro1) [7] |
|
 |
T19587 |
 |
|
|
| |
2.3.3.0.54 |
MYNN (OSZF, ZBTB31) [8] |
|
 |
T20033 |
 |
 |
|
| |
2.3.3.0.54.1 |
MYNN, isoform 1 |
|
 |
T20032 |
|
| |
2.3.3.0.54.2 |
MYNN, isoform 2 |
|
 |
T20034 |
|
| |
2.3.3.0.54.3 |
MYNN, isoform 3 |
|
 |
T20035 |
|
| |
2.3.3.0.55 |
ZBTB24 (ZNF450) [8] |
|
 |
T20221 |
 |
 |
|
| |
2.3.3.0.55.1 |
ZBTB24, isoform 1 |
|
 |
T20220 |
|
| |
2.3.3.0.55.2 |
ZBTB24, isoform 2 |
|
 |
T20223 |
|
| |
2.3.3.0.55.3 |
ZBTB24, isoform 3 |
|
 |
T20222 |
|
| |
2.3.3.0.57 |
ZFP92 [8] |
|
 |
T21421 |
 |
 |
|
| |
2.3.3.0.60 |
ZNF202 (ZKSCAN10, Zfp202) [8] |
GGGGTGGGGT |
 |
T |
 |
 |
|
| |
2.3.3.0.69 |
ZNF187 (ZSCAN26, SRE-ZBP, Zfp187) [8] |
|
 |
T16850 |
 |
 |
|
| |
2.3.3.0.69.1 |
ZNF187, isoform 1 |
|
 |
T16849 |
|
| |
2.3.3.0.69.2 |
ZNF187, isoform 2 |
|
 |
T16851 |
|
| |
2.3.3.0.70 |
ZSCAN22 (HKR2, ZNF50) [8] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.72 |
ZFP1 (ZNF475) [8] |
|
 |
T20280 |
 |
 |
|
| |
2.3.3.0.72.1 |
ZFP1, isoform 1 |
|
 |
T20279 |
|
| |
2.3.3.0.72.2 |
ZFP1, isoform 2 |
|
 |
T20281 |
|
| |
2.3.3.0.74 |
ZBTB16 (PLZF, ZNF145) [9] |
TAAAGTTTGATCTGTTC |
 |
T08243 |
 |
 |
|
| |
2.3.3.0.75 |
GTF3A (TFIIIA) [9] |
|
 |
T19312 |
 |
 |
 |
| |
2.3.3.0.76 |
ZFP64 [9] |
|
 |
T19406 |
 |
 |
 |
| |
2.3.3.0.76.1 |
ZFP64, isoform 1 |
|
 |
T19405 |
|
| |
2.3.3.0.76.2 |
ZFP64, isoform 2 |
|
 |
|
|
| |
2.3.3.0.76.3 |
ZFP64, isoform 3 [8] |
|
 |
|
|
| |
2.3.3.0.77 |
ZNF358 (Zfp358) [9] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.79 |
ZNF263 (FPM315, ZKSCAN12, Zfp263) [9] |
GGGAGGAGG |
 |
T22394 |
 |
 |
|
| |
2.3.3.0.81 |
ZNF192 (ZKSCAN8, LD5-1, Zfp192) [9] |
|
 |
T23327 |
 |
 |
|
| |
2.3.3.0.83 |
ZNF239 (HOK-2, MOK-2, Zfp239) [9] |
|
 |
T00510 |
 |
 |
|
| |
2.3.3.0.84 |
ZNF664 (ZFOC1, ZNF196, Zfp664) [9] |
|
 |
T20951 |
 |
 |
|
| |
2.3.3.0.92 |
GZF1 (ZBTB23, ZNF336) [10] |
TGCGCGTCTATA |
 |
T08231 |
 |
 |
|
| |
2.3.3.0.93 |
ZNF408 (PFM14, PRDM17, Zfp408) [10] |
|
 |
T |
 |
|
|
| |
2.3.3.0.94 |
ZNF648 (Zfp648) [10] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.95 |
ZNF768 (Zfp768) [10] |
|
 |
T21043 |
 |
 |
|
| |
2.3.3.0.101 |
ZNF483 (ZKSCAN16) [11] |
|
 |
T |
 |
|
|
| |
2.3.3.0.102 |
ZBTB48 (HKR3, ZNF855) [11] |
|
 |
T20247 |
 |
 |
 |
| |
2.3.3.0.102.1 |
ZBTB48, isoform 1 |
|
 |
T20246 |
|
| |
2.3.3.0.102.2 |
ZBTB48, isoform 2 |
|
 |
T20248 |
|
| |
2.3.3.0.103 |
ZNF275 (Zfp275) [11] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.114 |
ZNF354C (KID3) [11] |
CCACC |
 |
T20198 |
 |
 |
|
| |
2.3.3.0.120 |
ZSCAN12 (ZNF305, ZNF96) [11] |
|
 |
T21524 |
 |
 |
|
| |
2.3.3.0.123 |
ZBTB40 [12] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.125 |
ZNF454 (Zfp454) [12] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.148 |
ZNF329 (Zfp329) [12] |
|
 |
T20524 |
 |
 |
|
| |
2.3.3.0.149 |
ZFP90 (ZNF756) [13] |
|
 |
T20318 |
 |
 |
|
| |
2.3.3.0.150 |
ZNF317 (Zfp317) [13] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.152 |
MZF1 (ZNF42, ZSCAN6) [13] |
AGTGGGGA |
 |
T08517 |
 |
 |
|
| |
2.3.3.0.157 |
ZNF879 (Zfp879) [13] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.159 |
ZNF167 (ZKSCAN7, ZNF448, ZNF64, Zfp167) [13] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.160 |
ZNF250 (ZNF647, Zfp647) [13] |
|
 |
T19444 |
 |
 |
|
| |
2.3.3.0.162 |
ZFP3 (ZNF752) [13] |
|
 |
T20298 |
 |
 |
|
| |
2.3.3.0.166 |
ZNF311 (ZFP31, Zscan20) [14] |
|
 |
T |
 |
|
|
| |
2.3.3.0.166.1 |
ZNF311, isoform 1 |
|
 |
T |
|
| |
2.3.3.0.166.2 |
ZNF311, isoform 2 |
|
 |
T |
|
| |
2.3.3.0.167 |
ZNF551 (KOX23, Zfp551) [14] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.169 |
ZSCAN10 (ZNF206) [14] |
GCGCATGCGC |
 |
T18325 |
 |
 |
|
| |
2.3.3.0.169.1 |
ZSCAN10, isoform 1 |
|
 |
T18322 |
|
| |
2.3.3.0.169.2 |
ZSCAN10, isoform 2 |
|
 |
T18324 |
|
| |
2.3.3.0.169.3 |
ZSCAN10, isoform 3 |
|
 |
T18326 |
|
| |
2.3.3.0.174 |
ZNF287 (ZKSCAN13) [14] |
|
 |
T21498 |
 |
 |
|
| |
2.3.3.0.179 |
ZNF667 [15] |
|
 |
T20955 |
 |
 |
|
| |
2.3.3.0.192 |
ZFP28 [15] |
|
 |
T20285 |
 |
 |
 |
| |
2.3.3.0.192.1 |
ZFP28, isoform 1 |
|
 |
T20284 |
|
| |
2.3.3.0.192.2 |
ZFP28, isoform 2 |
|
 |
T20286 |
|
| |
2.3.3.0.193 |
ZNF182 (KOX14, ZNF21, Zfp182) [15] |
|
 |
T20399 |
 |
 |
|
| |
2.3.3.0.195 |
PRDM5 (PFM2) [16] |
GGAGAGCAGG |
 |
T20084 |
 |
 |
|
| |
2.3.3.0.196 |
RBAK (ZNF769) [16] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.197 |
ZNF189 (Zfp189) [16] |
|
 |
T |
 |
 |
|
| |
2.3.3.0.215 |
ZNF569 (Zfp74) [18] |
|
 |
T20803 |
 |
|
|
| |
2.3.3.0.219 |
ZNF271 (HZF7, ZNF-dp, ZNFEB, Zfp35) [19] |
|
 |
T00921 |
 |
 |
|
| |
2.3.3.0.220 |
ZNF184 (Zfp184) [19] |
|
 |
T20403 |
 |
|
|
| |
2.3.3.0.231 |
ZFP62 [23] |
|
 |
T |
 |
|
|
| |
2.3.3.0.231.1 |
ZFP62, isoform 1 |
|
 |
T |
|
| |
2.3.3.0.231.2 |
ZFP62, isoform 2 |
|
 |
T |
|
| |
2.3.3.0.231.3 |
ZFP62, isoform 3 |
|
 |
T |
|
| |
2.3.4 |
Family: Factors with multiple dispersed zinc fingers |
|
|
| |
2.3.4.1 |
Subfamily: ZNF417-like factors |
|
|
| |
2.3.4.2 |
Subfamily: ZNF219-like factors |
CCCCCA |
|
| |
2.3.4.2.1 |
ZNF219 (Zfp219) [2+1+2+1] |
|
 |
T |
 |
 |
|
| |
2.3.4.2.2 |
ZNF536 (Zfp536) [2+4+1+2] |
|
 |
T20746 |
 |
 |
|
| |
2.3.4.2.3 |
ZNF516 (Zfp516) [2+5+1+1+1] |
|
 |
T20711 |
 |
 |
|
| |
2.3.4.2.4 |
ZNF217 (ZABC1) [1+3+1+2] |
|
 |
T |
 |
 |
|
| |
2.3.4.3 |
Subfamily: Sal-like factors |
|
|
| |
2.3.4.3.1 |
SALL1 (Spalt-1, Sal-1, ZNF794) [2+3+2+2] |
|
 |
T19221 |
 |
 |
|
| |
2.3.4.3.2 |
SALL2 (Spalt-2, Sal-2, ZNF795) [2+3+2] |
|
 |
T19225 |
 |
 |
|
| |
2.3.4.3.3 |
SALL3 (Sal-3, ZNF796) [2+3+2+2] |
|
 |
T20099 |
 |
 |
|
| |
2.3.4.3.3.1 |
SALL3, isoform 1 [2+3+2+2] |
|
 |
T20098 |
|
| |
2.3.4.3.3.2 |
SALL3, isoform 2 [2+3] |
|
 |
T20100 |
|
| |
2.3.4.3.4 |
SALL4 (ZNF797) [2+3+2+2] |
|
 |
T19229 |
 |
 |
|
| |
2.3.4.3.4.1 |
SALL4, isoform 1 (Sall4a) [2+3+2+2] |
|
 |
T19228 |
|
| |
2.3.4.3.4.2 |
SALL4, isoform 2 (Sall4b) [2] |
|
 |
T26286 |
|
| |
2.3.4.3.4.3 |
SALL4, isoform 3 (Sall4c) [2] |
|
 |
T26287 |
|
| |
2.3.4.4 |
Subfamily: Ikaros |
TTGGGAAT |
|
| |
2.3.4.4.1 |
IKZF1 (IK1, IKAROS, Lyf-1, ZNFN1A1) [4+2] |
|
 |
T00479 |
 |
 |
|
| |
2.3.4.4.1.1 |
IKZF1, isoform I [3+2] |
|
 |
T01469 |
|
| |
2.3.4.4.1.2 |
IKZF1, isoform II [3+2] |
|
 |
T01470 |
|
| |
2.3.4.4.1.3 |
IKZF1, isoform III [3+2] |
|
 |
T01471 |
|
| |
2.3.4.4.1.4 |
IKZF1, isoform IV [1+2] |
|
 |
T01472 |
|
| |
2.3.4.4.1.5 |
IKZF1, isoform V [2] |
|
 |
T01473 |
|
| |
2.3.4.4.1.6 |
IKZF1, isoform VI |
|
 |
T19007 |
|
| |
2.3.4.4.2 |
IKZF2 (HELIOS, ZNFN1A2, Zfpn1a2) [4+2] |
|
 |
T06012 |
 |
 |
|
| |
2.3.4.4.2.1 |
IKZF2, isoform B |
|
 |
T11158 |
|
| |
2.3.4.4.2.2 |
IKZF2, isoform A [3+2] |
|
 |
T19008 |
|
| |
2.3.4.4.3 |
IKZF3 (Aiolos, ZNFN1A3) [4+2] |
|
 |
T05979 |
 |
 |
|
| |
2.3.4.4.4 |
IKZF4 (EOS, ZNFN1A4, Zfpn1a4) [4+2] |
|
 |
T19017 |
 |
 |
|
| |
2.3.4.4.4.1 |
IKZF4, isoform 1 |
|
 |
T19016 |
|
| |
2.3.4.4.4.2 |
IKZF4, isoform 2 |
|
 |
T26152 |
|
| |
2.3.4.4.4.3 |
IKZF4, isoform 3 |
|
 |
T26153 |
|
| |
2.3.4.4.4.4 |
IKZF4, isoform 4 |
|
 |
T26154 |
|
| |
2.3.4.4.5 |
IKZF5 (Pegasus, ZNFN1A5, Zfpn1a5) [3+2] |
|
 |
T19022 |
 |
 |
|
| |
2.3.4.4.5.1 |
IKZF5, isoform 1 |
|
 |
T19020 |
|
| |
2.3.4.4.5.2 |
IKZF5, isoform 2 |
|
 |
T19021 |
|
| |
2.3.4.4.5.3 |
IKZF5, isoform 3 |
|
 |
T26155 |
|
| |
2.3.4.5 |
Subfamily: HIV EP-factors |
GGGACTTTCC |
|
| |
2.3.4.5.1 |
HIV-EP1 (ZNF40, PRDII-BF1, MBP-1, CIRIP, GAAP) [2+1+2] |
|
 |
T00007 |
 |
 F F |
|
| |
2.3.4.5.2 |
HIV-EP2 (MBP-2, ZNF40B) [2+2] |
|
 |
T16993 |
 |
 |
|
| |
2.3.4.5.3 |
HIV-EP3 (KBP1, KRC, ZAS3) [2+1+2] |
|
 |
T02315 |
 |
 |
|
| |
2.3.4.6 |
Subfamily: ZBTB1-like factors |
|
|
| |
2.3.4.6.1 |
ZBTB1 [1+1+6] |
|
 |
T20252 |
 |
 |
|
| |
2.3.4.6.2 |
ZBTB2 (ZNF437) [1+3] |
|
 |
T |
 |
 |
|
| |
2.3.4.6.3 |
ZBTB25 (KUP, ZNF46) [1+1] |
|
 |
T |
 |
 |
|
| |
2.3.4.7 |
Subfamily: RLF-like factors |
|
|
| |
2.3.4.7.1 |
RLF [1+5+1+2+4+1] |
|
 F F |
T |
 |
|
|
| |
2.3.4.7.2 |
ZNF292 (Zfp292) [1+5+1+1+2+4+1] |
|
 |
T |
 |
 |
 |
| |
2.3.4.7.2.1 |
ZNF292, isoform 1 |
|
 |
T |
|
| |
2.3.4.7.2.2 |
ZNF292, isoform 2 |
|
 |
T |
|
| |
2.3.4.7.3 |
ZNF654 (Zfp654) [1+4] |
|
 |
T20939 |
 |
 |
|
| |
2.3.4.8 |
Subfamily: MAZ-like factors |
GGGGAGGG |
|
| |
2.3.4.8.1 |
MAZ (Zif87, ZNF801) [1+5] |
|
 |
T02303 |
 |
 |
|
| |
2.3.4.8.2 |
VEZF1 (DB1, ZNF161) [1+5] |
|
 |
T10444 |
 |
 |
|
| |
2.3.4.8.3 |
PATZ1 (RIAZ, ZBTB19, ZNF278, ZSG) [1+5+1] |
|
 |
T04796 |
 |
 |
 |
| |
2.3.4.9 |
Subfamily: ZNF532-like factors |
|
|
| |
2.3.4.9.1 |
ZNF532 (Zfp532) [1+6+3+2] |
|
 |
T20741 |
 |
 |
|
| |
2.3.4.9.1.1 |
ZNF532, isoform 1 |
|
 |
T20740 |
|
| |
2.3.4.9.1.2 |
ZNF532, isoform 2 |
|
 |
T20742 |
|
| |
2.3.4.9.2 |
ZNF592 (Zfp592) [2+6+3+2] |
|
 |
T19534 |
 |
 |
|
| |
2.3.4.9.3 |
ZNF687 (Zfp687) [1+5+2+2] |
|
 |
T20988 |
 |
 |
|
| |
2.3.4.9.3.1 |
ZNF687, isoform 1 |
|
 |
T20987 |
|
| |
2.3.4.9.3.2 |
ZNF687, isoform 2 [1+5+2] |
|
 |
T20989 |
|
| |
2.3.4.9.3.3 |
ZNF687, isoform 3 [1+5+2] |
|
 |
T20990 |
|
| |
2.3.4.10 |
Subfamily: ZNF512-factors |
|
|
| |
2.3.4.10.1 |
ZNF512 (Zfp512) [1+1+2] |
|
 |
T20697 |
 |
 |
 |
| |
2.3.4.10.2 |
ZNF512B (Zfp512b) [2+3+2] |
|
 F F |
T |
 |
 |
|
| |
2.3.4.11 |
Subfamily: ZNF658-factors |
|
|
| |
2.3.4.12 |
Subfamily: ZNF423-like factors |
GCACCCAAGGGTGC |
|
| |
2.3.4.12.1 |
ZNF423 (OAZ, Zfp423) [1+7+5+7+10] |
|
 |
T06599 |
 |
 |
|
| |
2.3.4.12.1.1 |
ZNF423, isoform 1 |
|
 |
T15869 |
|
| |
2.3.4.12.1.2 |
ZNF423, isoform 2 |
|
 |
T15870 |
|
| |
2.3.4.12.2 |
ZNF521 (EHZF, LIP3, Zpf521) [1+7+5+7+5+5] |
|
 |
T20719 |
 |
 |
|
| |
2.3.4.12.2.1 |
ZNF521, isoform 1 |
|
 |
T20718 |
|
| |
2.3.4.12.2.2 |
ZNF521, isoform 2 |
|
 |
T20720 |
|
| |
2.3.4.13 |
Subfamily: ZNF639-like factors |
|
|
| |
2.3.4.13.1 |
ZNF639 (ZASC1, Zfp639) [4+4] |
|
 |
T |
 |
 |
|
| |
2.3.4.13.2 |
ZNF711 (CMPX1, ZNF6, Zfp711) [1+10] |
|
 |
T |
 |
 |
|
| |
2.3.4.14 |
Subfamily: Evi-1-like factors |
ACAAGATAA |
|
| |
2.3.4.14.1 |
EVI-1 (MECOM) [7+3] |
|
 |
T00273 |
 |
 |
|
| |
2.3.4.14.1.1 |
EVI-1, isoform 1 |
|
 |
T10211 |
|
| |
2.3.4.14.1.2 |
EVI-1, isoform 2 |
|
 |
T25985 |
|
| |
2.3.4.14.2 |
PRDM16 (MEL1, PFM13) [7+3] |
|
 |
T27849 |
 |
 |
|
| |
2.3.4.14.2.1 |
PRDM16, isoform 1 |
|
 |
T27353 |
|
| |
2.3.4.14.2.2 |
PRDM16, isoform 2 (MEL1L) |
|
 |
T27927 |
|
| |
2.3.4.14.2.3 |
PRDM16, isoform 3 |
|
 |
T27354 |
|
| |
2.3.4.15 |
Subfamily: BCL11 |
|
|
| |
2.3.4.15.1 |
BCL11A (CTIP1, EVI9, ZNF856) [1+2] |
|
 |
T27831 |
 |
 |
|
| |
2.3.4.15.1.1 |
BCL11A, isoform 1 (a) [1+2] |
|
 |
T27221 |
|
| |
2.3.4.15.1.2 |
BCL11A, isoform 2 (b) [2] |
|
 |
T27873 |
|
| |
2.3.4.15.1.3 |
BCL11A, isoform 3 (c) [1] |
|
 |
T27874 |
|
| |
2.3.4.15.1.4 |
BCL11A, isoform 4 [2] |
|
 |
T27875 |
|
| |
2.3.4.15.1.5 |
BCL11A, isoform 5 [1] |
|
 |
T27222 |
|
| |
2.3.4.15.1.6 |
BCL11A, isoform 6 [1] |
|
 |
T27876 |
|
| |
2.3.4.15.1.7 |
BCL11A, isoform 7 [0] |
|
 |
T27877 |
|
| |
2.3.4.15.1.8 |
BCL11A, isoform 8 [2] |
|
 |
T27878 |
|
| |
2.3.4.15.2 |
BCL11B (CTIP2, RIT1) [1+2+3] |
|
 |
T |
 |
 |
|
| |
2.3.4.15.2.1 |
BCL11B, isoform 1 (α) |
|
 |
T |
|
| |
2.3.4.15.2.2 |
BCL11B, isoform 2 (β) |
|
 |
T |
|
| |
2.3.4.15.2.3 |
BCL11B, isoform 3 (γ) |
|
 |
T |
|
| |
2.3.4.15.3 |
ZNF296 (ZNF342) [1+2+3] |
|
 |
T |
 |
 |
|
| |
2.3.4.16 |
Subfamily: Insulinoma-associated proteins |
|
|
| |
2.3.4.16.1 |
INSM1 (IA-1) [2+1+2] |
|
 |
T16447 |
 |
|
 |
| |
2.3.4.16.2 |
INSM2 (IA-6) [2+3] |
|
 |
T21510 |
 |
 |
|
| |
2.3.4.17 |
Subfamily: Hypermethylated in Cancer proteins |
GGGTGCCCGG |
|
| |
2.3.4.17.1 |
HIC1 (ZBTB29) [1+4] |
 |
 |
T06430 |
 |
 F F |
|
| |
2.3.4.17.2 |
HIC2 (HRG22, ZBTB30) [1+4] |
 |
 |
T18974 |
 |
 |
|
| |
2.3.4.17.2.1 |
HIC2, isoform 1 |
|
 |
T18976 |
|
| |
2.3.4.17.2.2 |
HIC2, isoform 2 [1+1] |
|
 |
T18975 |
|
| |
2.3.4.18 |
Subfamily: ZNF518 |
|
|
| |
2.3.4.18.1 |
ZNF518A (Zfp518a) [4+1] |
|
 |
T |
 |
 |
|
| |
2.3.4.18.2 |
ZNF518B (Zfp518b) [2+1] |
|
 |
T21344 |
 |
 |
|
| |
2.3.4.19 |
Subfamily: Basonuclin |
|
|
| |
2.3.4.19.1 |
BNC1 [2+2+2] |
|
 |
T05192 |
 |
 |
|
| |
2.3.4.19.2 |
BNC2 [1+1+2] |
|
 |
T |
 |
 |
|
| |
2.3.4.19.2.1 |
BNC2, isoform 1 |
|
 |
T |
|
| |
2.3.4.19.2.2 |
BNC2, isoform 2 |
|
 |
T |
|
| |
2.3.4.20 |
Subfamily: TRERF1-like factors |
|
|
| |
2.3.4.20.1 |
TRERF1 (BCAR2, RAPA, TREP132) = 3.5.1.3.9 [1+1+1] |
|
 |
T16489 |
 |
 |
|
| |
2.3.4.20.1.1 |
TRERF1, isoform 1 |
|
 |
T16488 |
|
| |
2.3.4.20.1.2 |
TRERF1, isoform 2 |
|
 |
T16490 |
|
| |
2.3.4.20.1.3 |
TRERF1, isoform 3 [1] |
|
 |
T16491 |
|
| |
2.3.4.20.1.4 |
TRERF1, isoform 4 |
|
 |
T16492 |
|
| |
2.3.4.20.2 |
ZNF541 (Ship1, Zfp541) [3+1+1] = 3.5.1.3.10 |
|
 |
T |
 |
 |
|
| |
2.3.4.20.2.1 |
ZNF541, isoform 1 |
|
 |
T |
|
| |
2.3.4.20.2.2 |
ZNF541, isoform 2 |
|
 |
T |
|
| |
2.3.4.21 |
Subfamily: HINFP-like factors |
CGGACGTT |
|
| |
2.3.4.21.1 |
HINFP (MIZF, ZNF743) [1+8] |
 |
 |
T |
 |
 |
|
| |
2.3.4.21.2 |
ZFAT (ZFAT1, ZNF406, Zfp406) [1+1+7+10] |
|
 |
T20270 |
 |
 |
 |
| |
2.3.4.21.2.1 |
ZFAT, isoform 1 |
|
 |
T20269 |
|
| |
2.3.4.21.2.2 |
ZFAT, isoform 2 |
|
 |
T20271 |
|
| |
2.3.4.22 |
Subfamily: ZNF526-like factors |
|
|
| |
2.3.4.22.1 |
ZNF526 (Zfp526) [1+2+1+9] |
|
 |
T20728 |
 |
 |
|
| |
2.3.4.22.1.1 |
ZNF526, isoform 1 |
|
 |
T20727 |
|
| |
2.3.4.22.1.2 |
ZNF526, isoform 2 [1+2+1+4] |
|
 |
T20729 |
|
| |
2.3.4.22.2 |
ZNF574 [3+1+4+8+4] |
|
 |
T20819 |
 |
 |
|
| |
2.3.4.23 |
Subfamily: ZNF319-like factors |
|
|
| |
2.3.4.23.1 |
ZNF319 (Zfp319) [3+13] |
|
 |
T20516 |
 |
 |
|
| |
2.3.4.23.2 |
ZNF786 (Zfp786) [2+14] |
|
 |
T21088 |
 |
 |
|
| |
2.3.4.24 |
Subfamily: ZNF134-like factors |
|
|
| |
2.3.4.25 |
Subfamily: ZNF37A-like factors |
|
|
| |
2.3.4.25.3 |
ZNF248 (Zfp248) [1+7] |
|
 |
T |
 |
 |
|
| |
2.3.4.25.4 |
ZNF334 (Zfp334) [9+5] |
|
 |
T |
 |
 |
|
| |
2.3.4.25.5 |
ZNF382 (KS1, Zfp382) [1+9] |
|
 |
T20560 |
 |
 |
|
| |
2.3.4.0 |
Subfamily: unclassified |
|
|
| |
2.3.4.0.1 |
CASZ1 (CST, SRG, ZNF693) [3+1+1+3] |
|
 |
T19862 |
 |
 |
|
| |
2.3.4.0.1.1 |
CASZ1, isoform 1 |
|
 |
T19861 |
|
| |
2.3.4.0.1.2 |
CASZ1, isoform 2 [3+1] |
|
 |
T19863 |
|
| |
2.3.4.0.2 |
ZNF335 (NIF-1, Zfp335) [1+8+4] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.3 |
PRDM8 (PFM5) [1+2] |
|
 |
T20089 |
 |
 |
|
| |
2.3.4.0.4 |
WIZ (ZNF803) [4+2+1+1+1+1+1] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.4.1 |
WIZ, isoform L |
|
 |
T |
|
| |
2.3.4.0.4.2 |
WIZ, isoform S [1+1+1+1+1+1] |
|
 |
T |
|
| |
2.3.4.0.4.3 |
WIZ, isoform 2 [1+1+1+1+1] |
|
 |
T |
|
| |
2.3.4.0.4.4 |
WIZ, isoform 3 [4] |
|
 |
T |
|
| |
2.3.4.0.5 |
ZNF469 [1+1+3] |
|
 F F |
T |
 |
 |
|
| |
2.3.4.0.6 |
ZFPM1 (FOG1, ZFN89A) = 2.7.2.0.1 [3+1] |
|
 |
T02334 |
 |
 |
|
| |
2.3.4.0.7 |
ZBTB4 (KAISO-L1) [1+3+2] |
|
 |
T |
 |
 |
 |
| |
2.3.4.0.8 |
ZBTB38 [2+3+5] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.9 |
ZBTB39 [2+3+3] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.10 |
ZNF295 (ZBTB21) [2+1+2+2+1] |
|
 |
T |
 |
|
 |
| |
2.3.4.0.11 |
ZNF827 [3+4+2] |
|
 |
T21121 |
 |
|
|
| |
2.3.4.0.11.1 |
ZNF827, isoform 1 |
|
 |
T21120 |
|
| |
2.3.4.0.11.2 |
ZNF827, isoform 2 [2] |
|
 |
T21122 |
|
| |
2.3.4.0.12 |
PRDM13 (PFM10) [1+3] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.12.1 |
PRDM13, isoform 1 |
|
 |
T |
|
| |
2.3.4.0.12.2 |
PRDM13, isoform 2 |
|
 |
T |
|
| |
2.3.4.0.13 |
ZNF646 (PFM10, Zfp646) [1+3] |
|
 |
T |
 |
 F F |
|
| |
2.3.4.0.14 |
ZNF579 (Zfp579) [3+2+3] |
|
 |
T20833 |
 |
 |
|
| |
2.3.4.0.15 |
RREB1 (FINB, Zep-1, LZ321) [3+2+1+5+1+1+2] |
CCCCAAACACCCCC |
 |
T17652 |
 |
 |
|
| |
2.3.4.0.15.1 |
RREB1, isoform 1 |
|
 |
T17651 |
|
| |
2.3.4.0.15.2 |
RREB1, isoform 2 [3+2+1+5+1+1+2]] |
|
 |
T17653 |
|
| |
2.3.4.0.15.3 |
RREB1, isoform 3 [3+2+1+5+2+1+2] |
|
 |
T17654 |
|
| |
2.3.4.0.15.4 |
RREB1, isoform 4 [3+2+1+5+1] |
|
 |
T17655 |
|
| |
2.3.4.0.16 |
PRDM2 (G3B, MTB-ZF, MTE-BP, KMT8, RIZ) [2+1+3+1+1] |
GTCATATGAC |
 |
T |
 |
 |
|
| |
2.3.4.0.17 |
ZNF800 (Zfp800) [1+3+2+1] |
|
 |
T21110 |
 |
 |
|
| |
2.3.4.0.18 |
ZNF618 [3+1] |
|
 |
T21454 |
 |
 F F |
|
| |
2.3.4.0.18.1 |
ZNF618, isoform 1 |
|
 |
T21453 |
|
| |
2.3.4.0.18.2 |
ZNF618, isoform 2 [3] |
|
 |
T21455 |
|
| |
2.3.4.0.19 |
ZNF277 (NRIF4, ZNF277P, Zfp277) [1+1] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.20 |
TRPS1 (GC79) = 2.2.1.2.13 [1+1+3+2] |
|
 |
T19349 |
 |
 |
|
| |
2.3.4.0.21 |
ZNF451 (COASTER, Zfp451) [1+3+2+3+2] |
|
 |
T19504 |
 |
 |
|
| |
2.3.4.0.22 |
ZNF438 [3+1] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.23 |
PEG3 (ZSCAN24) [4+1+5+2] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.23.1 |
PEG3, isoform 1 |
|
 |
|
|
| |
2.3.4.0.23.2 |
PEG3, isoform 2 [2] |
|
 |
T22527 |
|
| |
2.3.4.0.24 |
ZNF507 (Zfp507) [2+1+3+2+1] |
|
 |
T20688 |
 |
 |
|
| |
2.3.4.0.25 |
ZNF236 (Zfp236) [9+4+4+4+4+5] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.26 |
ZNF770 (Zfp770) [3+3+1+2+2] |
|
 |
T21047 |
 |
 |
|
| |
2.3.4.0.27 |
REST (NRSF, XBR) [1+7+1] |
TTCAGCACCACGGACAGCGCC |
 |
T05934 |
 |
 |
|
| |
2.3.4.0.27.1 |
REST, isoform 1 |
|
 |
T09307 |
|
| |
2.3.4.0.27.2 |
REST, isoform 2 (N16, REST4) [1+4] |
|
 |
T10486 |
|
| |
2.3.4.0.27.3 |
REST, isoform 3 (N28) [1+4] |
|
 |
T26267 |
|
| |
2.3.4.0.28 |
ZFP57 (ZNF698) [4+3] |
TTCAGCACCACGGACAGCGCC |
 |
T20308 |
 |
 |
 |
| |
2.3.4.0.28.1 |
ZFP57, isoform 1 |
|
 |
T20307 |
|
| |
2.3.4.0.28.2 |
ZFP57, isoform 2 |
|
 |
T20309 |
|
| |
2.3.4.0.29 |
ZNF8 (HF.18, Zfp128) [6+1] |
|
 |
T22794 |
 |
 |
|
| |
2.3.4.0.30 |
ZNF597 (Zfp597) [4+3] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.31 |
PRDM4 (PFM1) [1+5] |
 |
 |
T18699 |
 |
 |
|
| |
2.3.4.0.32 |
PRDM15 (ZNF298) [1+15] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.33 |
ZBTB41 (FRBZ1) [1+13] |
|
 |
T20243 |
 |
 |
|
| |
2.3.4.0.34 |
PRDM10 (PFM7, TRIS) [1+9] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.34.1 |
PRDM10, isoform 1 |
|
 |
|
|
| |
2.3.4.0.34.2 |
PRDM10, isoform 2 [0] |
|
 |
|
|
| |
2.3.4.0.35 |
ZNF341 (Zfp341) [1+2+9] |
|
 |
T |
 |
 F F |
|
| |
2.3.4.0.36 |
ZNF513 (Zfp513) [3+5] |
|
 |
T20703 |
 |
 |
|
| |
2.3.4.0.36.1 |
ZNF513, isoform 1 |
|
 |
T20702 |
|
| |
2.3.4.0.36.2 |
ZNF513, isoform 2 |
|
 |
T20704 |
|
| |
2.3.4.0.37 |
ZNF142 (Zfp142) [14+17] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.38 |
FIZ1 (ZNF798) [4+2+5] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.39 |
ZNF628 (Zfp628) [7+2+6] |
CAAGGTTGGTTGC |
 |
T08324 |
 |
 |
|
| |
2.3.4.0.39.1 |
ZNF628, isoform 1 |
|
 |
T08319 |
|
| |
2.3.4.0.39.2 |
ZNF628, isoform 2 [7] |
|
 |
T26369 |
|
| |
2.3.4.0.40 |
ZNF784 (Zfp784) [3+3] |
 |
 |
T21081 |
 |
 |
|
| |
2.3.4.0.41 |
ZNF697 (Zfp697) [1+10] |
|
 |
T21002 |
 |
 |
|
| |
2.3.4.0.42 |
ZKSCAN5 (ZFP95) [4+9] |
|
 |
T21526 |
 |
 |
|
| |
2.3.4.0.43 |
ZNF316 (Zfp316) [6+9] |
|
 |
T20264 |
 |
 |
|
| |
2.3.4.0.44 |
ZNF775 (Zfp775) [4+4+3] |
|
 |
T21063 |
 |
 |
|
| |
2.3.4.0.45 |
ZNF467 (Zfp467) [7+5] |
|
 |
T19511 |
 |
 |
|
| |
2.3.4.0.46 |
ZNF787 (TTF-I-IP20, Zfp787) [5+2] |
|
 |
T21090 |
 |
 |
|
| |
2.3.4.0.48 |
ZNF48 (ZNF553, Zfp48, Zfp553) [8+2+2] |
|
 |
T21192 |
 |
 |
|
| |
2.3.4.0.49 |
ZNF629 (ZNF65, Zfp629) [15+3+1] |
|
 |
T20905 |
 |
 |
|
| |
2.3.4.0.50 |
ZNF668 (Zfp668) [13+3] |
|
 |
T20959 |
 |
 |
|
| |
2.3.4.0.53 |
ZNF445 (ZKSCAN15, ZNF168, Zfp445) [12+2] |
|
 |
T20627 |
 |
 |
|
| |
2.3.4.0.56 |
ZSCAN20 (KOX29, ZNF31, ZNF360) [4+6] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.56.1 |
ZSCAN20, isoform 1 |
|
 |
T |
|
| |
2.3.4.0.56.2 |
ZSCAN20, isoform 2 |
|
 |
T |
|
| |
2.3.4.0.64 |
ZNF426 (Zfp426) [1+11] |
|
 |
T19487 |
 |
 |
|
| |
2.3.4.0.65 |
ZFP112 (ZNF112, ZNF228) [1+16] |
|
 |
T21386 |
 |
 |
|
| |
2.3.4.0.65.1 |
ZFP112, isoform 1 |
|
 |
T21385 |
|
| |
2.3.4.0.65.2 |
ZFP112, isoform 2 |
|
 |
T21387 |
|
| |
2.3.4.0.67 |
ZNF606 (ZNF328, Zfp606) [2+14] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.71 |
PRDM9 (PFM6) [1+12] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.71.1 |
PRDM9, isoform 1 (Meisetz) |
|
 |
|
|
| |
2.3.4.0.71.2 |
PRDM9, isoform 2 (Meisetz-S1) [0] |
|
 |
|
|
| |
2.3.4.0.71.3 |
PRDM9, isoform 3 (Meisetz-S2) [0] |
|
 |
|
|
| |
2.3.4.0.71.4 |
PRDM9, isoform 4 [0] |
|
 |
|
|
| |
2.3.4.0.72 |
ZBTB17 (MIZ1, ZNF151, ZNF60) [12+1] |
|
 |
T17704 |
 |
 |
|
| |
2.3.4.0.73 |
ZNF407 (Zfp407) [3+3+1+2+2+11] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.74 |
E4F1 (p120E4F, p50E4F) [3+6] |
TACGTCAC |
 |
T02287 |
 |
 |
|
| |
2.3.4.0.74.1 |
E4F1, isoform 1 |
|
 |
T14957 |
|
| |
2.3.4.0.74.2 |
E4F1, isoform 2 |
|
 |
T26091 |
|
| |
2.3.4.0.74.3 |
E4F1, isoform 3 |
|
 |
T26092 |
|
| |
2.3.4.0.74.4 |
E4F1, isoform 4 [3+1] |
|
 |
T26093 |
|
| |
2.3.4.0.74.5 |
E4F1, isoform 5 [2+6] |
|
 |
T26094 |
|
| |
2.3.4.0.75 |
ZNF462 (Zfp462) [1+2+2+1+3+1+1+3+2+1+4+5+1] |
|
 |
T |
 |
 |
 |
| |
2.3.4.0.76 |
ZNF644 (ZEP2, Zfp644) [5+1+1] |
|
 |
T |
 |
 |
|
| |
2.3.4.0.77 |
ADNP (ADNP1) = 3.1.8.1.1 [4+3+2] |
|
 |
T21522 |
 |
 |
|
| |
2.3.4.0.78 |
ADNP2 (ZNF508) = 3.1.8.1.2 [1+1+1+1] |
|
 |
T19732 |
 |
 |
|
| |
2.3.5 |
Family: BED zinc finger factors |
TGTCGCGACA |
|
| |
2.3.5.0.3 |
ZBED3 |
|
 |
T |
 |
 |
|
| |
2.3.5.0.4 |
ZBED4 |
|
 |
T |
 |
 |
|
| |
2.3.5.0.6 |
ZBED6 (MGR) |
|
 |
T17368 |
 |
|
|
| |
2.3.5.0.6.1 |
ZBED6, isoform 1 (a) |
|
 |
T17346 |
|
| |
2.3.5.0.6.2 |
ZBED6, isoform 2 (b) |
|
 |
T17365 |
|
| |
2.4 |
Class: C6 zinc cluster factors |
 |
| |
| |
2.5 |
Class: DM-type intertwined zinc finger factors |
 |
 |
| |
2.5.1 |
Family: DMRT |
TGATACATTGTTGC |
|
| |
2.5.1.0.1 |
DMRT1 |
|
 |
T10713 |
 |
 |
|
| |
2.5.1.0.1.1 |
DMRT1, isoform 1 (a) |
|
 |
T10712 |
|
| |
2.5.1.0.1.2 |
DMRT1, isoform 2 (b) |
|
 |
T26413 |
|
| |
2.5.1.0.1.3 |
DMRT1, isoform 3 (d) |
|
 |
T26414 |
|
| |
2.5.1.0.1.4 |
DMRT1, isoform 4 (c) |
|
 |
T26415 |
|
| |
2.5.1.0.2 |
DMRT2 |
|
 |
T10722 |
 |
 |
|
| |
2.5.1.0.3 |
DMRT3 |
|
 |
T10724 |
 |
 |
|
| |
2.5.1.0.4 |
DMRTA1 (DMRT4) |
|
 |
T10728 |
 |
 |
|
| |
2.5.1.0.5 |
DMRTA2 (DMRT5) |
|
 |
T10732 |
 |
 |
|
| |
2.5.1.0.6 |
DMRTB1 |
|
 |
T10730 |
 |
 |
|
| |
2.5.1.0.7 |
DMRTC2 |
|
 |
T10726 |
 |
 |
|
| |
2.5.1.0.8 |
DMRTC1a (no DM domain!) |
|
 |
T |
 |
 |
|
| |
2.5.1.0.8.1 |
DMRTC1a, isoform 1 (c) |
|
 |
T |
|
| |
2.5.1.0.8.2 |
DMRTC1a, isoform 2 (b) |
|
 |
T |
|
| |
2.5.1.0.8.3 |
DMRTC1a, isoform 3 (a) |
|
 |
T |
|
| |
2.5.1.0.9 |
DMRTC1b (no DM domain!) |
|
 |
T |
 |
 |
|
| |
2.5.1.0.10 |
DMRTC1c (no DM domain!) |
|
 |
T |
 |
|
|
| |
2.6 |
Class: CXXC zinc finger factors |
 |
 |
| |
2.6.1 |
Family: CpG-binding proteins |
|
|
| |
2.6.1.0.1 |
CpG-binding protein (CXXC1, CFP1, CGBP, PCCX1, PHF18) |
|
 |
T19901 |
 |
 |
|
| |
2.6.1.0.2 |
MBD1 (CXXC3, PCM1) |
|
 |
T |
 |
 |
|
| |
2.6.1.0.2.1 |
MBD1, isoform 1 (a) |
|
 |
T |
|
| |
2.6.1.0.2.2 |
MBD1, isoform 2 (b) |
|
 |
T |
|
| |
2.6.1.0.2.3 |
MBD1, isoform 3 (d) |
|
 |
T |
|
| |
2.6.1.0.2.4 |
MBD1, isoform 4 |
|
 |
T |
|
| |
2.6.1.0.2.5 |
MBD1, isoform 5 |
|
 |
T |
|
| |
2.6.1.0.3 |
TET1 (CXXC6, LCX) |
|
 |
T |
 |
 |
|
| |
2.6.1.0.4 |
MLL (CXXC7, ALL1, HRX, HTRX, KMT2A, MLL1, TRX1) |
|
 |
T27835 |
 |
 |
 |
| |
2.6.1.0.4.1 |
MLL, isoform 1 |
|
 |
T27239 |
|
| |
2.6.1.0.4.2 |
MLL, isoform 14P-18B |
|
 |
T27889 |
|
| |
2.6.1.0.5 |
KDM2A (CXXC8, FBL7, FBXL11, JHDM1A) |
|
 |
T27843 |
 |
 |
|
| |
2.6.1.0.5.1 |
KDM2A, isoform 1 |
|
 |
T27346 |
|
| |
2.6.1.0.5.2 |
KDM2A, isoform 2 |
|
 |
T27920 |
|
| |
2.6.1.0.5.3 |
KDM2A, isoform 3 |
|
 |
T27921 |
|
| |
2.6.1.0.6 |
DNMT1 (CXXC9, AIM, DNMT) |
|
 |
T27826 |
 |
 |
|
| |
2.6.1.0.6.1 |
DNMT1, isoform 1 (long) |
|
 |
T27185 |
|
| |
2.6.1.0.6.2 |
DNMT1, isoform 2 (short) |
|
 |
T27865 |
|
| |
2.6.1.0.7 |
MLL2 (HRX2, KMT2B, WBP7, MLL4, TRX2) |
|
 |
T19083 |
 |
 |
|
| |
2.7 |
Class: C2HC zinc finger factors |
 |
 |
| |
2.7.1 |
Family: Myelin transcription factor-related proteins |
AAGTT |
|
| |
2.7.1.0.1 |
MYT1 (MyT1, MTF1, MYTI, PLPB1) [7] |
|
 |
T22947 |
 |
 |
 |
| |
2.7.1.0.1.1 |
MYT1, isoform 1 (NZF-2b) |
|
 |
T25788 |
|
| |
2.7.1.0.1.2 |
MYT1, isoform 2 (NZF-2a) [6] |
|
 |
T25789 |
|
| |
2.7.1.0.1.3 |
MYT1, isoform 3 [6] |
|
 |
T26205 |
|
| |
2.7.1.0.2 |
MYT1L (MyT1L, NZF-1) [6] |
|
 |
T27828 |
 |
 |
|
| |
2.7.1.0.2.1 |
MYT1L, isoform 1 |
|
 |
T27211 |
|
| |
2.7.1.0.2.2 |
MYT1L, isoform 2 |
|
 |
T27212 |
|
| |
2.7.1.0.2.3 |
MYT1L, isoform 3 [0] |
|
 |
|
|
| |
2.7.1.0.2.4 |
MYT1L, isoform 4 [2] |
|
 |
|
|
| |
2.7.1.0.3 |
ST18 (ZNF387, NZF-3) [6] |
|
 |
T |
 |
 |
|
| |
2.7.2 |
Family: Friend of GATA proteins |
|
|
| |
2.7.2.0.1 |
FOG1 (ZFPM1, ZFN89A) [5] = 2.3.4.0.6 |
|
 |
T02334 |
 |
 |
|
| |
2.7.2.0.2 |
FOG2 (ZFPM2, ZFN89B) [5] = 2.3.2.4.6 |
|
 |
T18922 |
 |
 F F |
|
| |
2.7.3 |
Family: Histone acetyltransferases with C2HC zinc finger |
|
|
| |
2.7.3.0.1 |
MYST1 (KAT8, TIP60 protein 1) [1] |
|
 |
T27842 |
 |
 |
|
| |
2.7.3.0.1.1 |
MYST1, isoform 1 |
|
 |
T27345 |
|
| |
2.7.3.0.1.2 |
MYST1, isoform 2 [0] |
|
 |
T27919 |
|
| |
2.7.3.0.2 |
MYST2 (KAT7, TIP60 protein 2, HBO1, HBOa) [1] |
|
 |
T |
 |
 |
|
| |
2.7.3.0.2.1 |
MYST2, isoform 1 |
|
 |
T |
|
| |
2.7.3.0.2.2 |
MYST2, isoform 2 |
|
 |
T |
|
| |
2.7.3.0.2.3 |
MYST2, isoform 3 |
|
 |
T |
|
| |
2.7.3.0.2.4 |
MYST2, isoform 4 |
|
 |
T |
|
| |
2.7.3.0.2.5 |
MYST2, isoform 5 |
|
 |
T |
|
| |
2.7.3.0.3 |
MYST3 (KAT6A, TIP60 protein 3, RUNXBP2, ZNF220, MOZ) [1] |
|
 |
T |
 |
 |
|
| |
2.7.3.0.4 |
MYST4 (KAT6B, TIP60 protein 4, MORF, MOZ2) [1] |
|
 |
T |
 |
|
|
| |
2.7.3.0.4.1 |
MYST4, isoform 1 |
|
 |
T |
|
| |
2.7.3.0.4.2 |
MYST4, isoform 2 |
|
 |
T |
|
| |
2.7.3.0.5 |
KAT5 (HTATIP, TIP60) [1] |
|
 |
T27824 |
 |
 |
|
| |
2.7.3.0.5.1 |
KAT5, isoform 1 |
|
 |
T27282 |
|
| |
2.7.3.0.5.2 |
KAT5, isoform 2 |
|
 |
T27283 |
|
| |
2.7.3.0.5.3 |
KAT5, isoform 3 |
|
 |
T27284 |
|
| |
2.7.3.0.5.4 |
KAT5, isoform 4 |
|
 |
T27944 |
|
| |
2.7.4 |
Family: Lethal(3)malignant brain tumor-related proteins |
|
|
| |
2.7.4.0.1 |
L3MBTL1 (L3MBT, L3MBTL) [1] |
|
 |
T |
 |
 |
|
| |
2.7.5 |
Family: LYAR-related proteins |
|
|
| |
2.7.5.0.1 |
LYAR (PNAS-5) [2] |
|
 |
T |
 |
 |
|
| |
2.8 |
Class: C3H zinc finger factors |
 |
 |
| |
2.8.1 |
Family: ZC3H8-related factors |
|
|
| |
2.8.1.0.1 |
ZC3H8 (ZC3HDC8, FLIZ1) [3] |
|
 |
T |
 |
 |
|
| |
2.8.1.0.2 |
ZC3H4 [3] |
|
 |
T |
 |
 |
|
| |
2.8.1.0.2.1 |
ZC3H4, isoform 1 |
|
 |
T |
|
| |
2.8.1.0.2.2 |
ZC3H4, isoform 2 |
|
 |
T |
|
| |
2.8.1.0.3 |
ZC3H6 (ZC3HDC6) [3] |
|
 |
T |
 |
 |
|
| |
2.8.1.0.3.1 |
ZC3H6, isoform 1 |
|
 |
|
|
| |
2.8.1.0.3.2 |
ZC3H6, isoform 2 [0] |
|
 |
|
|
| |
2.8.2 |
Family: ZGPAT-related factors |
|
|
| |
2.8.2.0.1 |
ZGPAT (GPATC6, GPATCH6, ZC3H9, ZC3HDC9, ZIP) [1] |
|
 |
T27855 |
 |
 |
|
| |
2.8.3 |
Family: RC3H-related factors |
|
|
| |
2.8.3.0.1 |
RC3H2 (Roquin-2, MNAB, RNF164) [1] |
|
 |
T |
 |
 |
|
| |
2.8.4 |
Family: CNOT4-related factors |
|
|
| |
2.8.4.0.1 |
CNOT4 (NOT4) [1] |
|
 |
T27838 |
 |
 |
|
| |
2.8.4.0.1.1 |
CNOT4, isoform 1 |
|
 |
T27257 |
|
| |
2.8.4.0.1.2 |
CNOT4, isoform 2 |
|
 |
T27258 |
|
| |
2.8.4.0.1.3 |
CNOT4, isoform 3 |
|
 |
T27940 |
|
| |
2.9 |
Class: C2CH THAP-type zinc finger factors |
 |
 |
| |
2.9.1 |
Family: THAP-related factors |
TTGCCCGTACT |
|
| |
2.9.1.0.1 |
THAP1 |
|
 |
T |
 |
 |
 |
| |
2.9.1.0.2 |
THAP2 |
|
 |
T |
 |
 |
|
| |
2.9.1.0.3 |
THAP3 |
|
 |
T |
 |
 |
|
| |
2.9.1.0.4 |
THAP4 (CGI-36, PP238) |
|
 |
T |
 |
 |
|
| |
2.9.1.0.4.1 |
THAP4, isoform 1 |
|
 |
T |
|
| |
2.9.1.0.4.2 |
THAP4, isoform 2 |
|
 |
T |
|
| |
2.9.1.0.7 |
THAP7 |
|
 |
T |
 |
 |
|
| |
2.9.1.0.11 |
THAP11 (HRIHFB2206) |
|
 |
T |
 |
 |
|
| |
2.9.1.0.12 |
PRKRIR (P52RIPK, DAP4, THAP0) |
|
 |
T |
 |
 |
|
|
| 3 |
Superclass: Helix-turn-helix domains |
 |
| |
3.1 |
Class: Homeo domain factors |
 |
 |
| |
3.1.1 |
Family: HOX-related factors |
|
|
| |
3.1.1.1 |
Subfamily: HOX1 |
TAATTA |
|
| |
3.1.1.1.1 |
Hox-A1 (HOX1F, ERA-1) |
 |
 |
T01696 |
 |
 |
|
| |
3.1.1.1.1.1 |
Hox-A1, isoform 1 (ERA-1-993) |
|
 |
|
|
| |
3.1.1.1.1.2 |
Hox-A1, isoform 2 (ERA-1-399) |
|
 |
|
|
| |
3.1.1.1.2 |
Hox-B1 (HOX2I) |
|
 |
T01720 |
 |
 |
 |
| |
3.1.1.1.3 |
Hox-D1 (HOX4G) |
|
 |
T01750 |
 |
 |
|
| |
3.1.1.2 |
Subfamily: HOX2 |
TAATTA |
|
| |
3.1.1.2.1 |
Hox-A2 (HOX1K) |
 |
 |
T13907 |
 |
 |
|
| |
3.1.1.2.2 |
Hox-B2 (HOX2H) |
 |
 |
T19758 |
 |
 |
|
| |
3.1.1.3 |
Subfamily: HOX3 |
TAATTA |
|
| |
3.1.1.3.1 |
Hox-A3 (HOX1E) |
|
 |
T00378 |
 |
 |
|
| |
3.1.1.3.2 |
Hox-B3 (HOX2G) |
 |
 |
T01724 |
 |
 |
|
| |
3.1.1.3.3 |
Hox-D3 (HOX4A) |
 |
 |
T03339 |
 |
 |
|
| |
3.1.1.4 |
Subfamily: HOX4 |
TTAATTAA |
|
| |
3.1.1.4.1 |
Hox-A4 (HOX1D) |
|
 |
T01701 |
 |
 F F |
|
| |
3.1.1.4.2 |
Hox-B4 (HOX2F) |
|
 |
T01728 |
 |
 |
|
| |
3.1.1.4.3 |
Hox-C4 (HOX3E) |
|
 |
T03340 |
 |
 |
|
| |
3.1.1.4.4 |
Hox-D4 (HOX4B) |
|
 |
T |
 |
 |
|
| |
3.1.1.5 |
Subfamily: HOX5 |
TAATTA |
|
| |
3.1.1.5.1 |
Hox-A5 (HOX1C) |
|
 |
T00377 |
 |
 |
|
| |
3.1.1.5.2 |
Hox-B5 (HOX2A) |
 |
 |
T13933 |
 |
 |
|
| |
3.1.1.5.3 |
Hox-C5 (HOX3D) |
|
 |
T03351 |
 |
 |
|
| |
3.1.1.6 |
Subfamily: HOX6-7 |
TAATTA |
|
| |
3.1.1.6.1 |
Hox-A6 (HOX1B) |
|
 |
T03338 |
 |
 |
|
| |
3.1.1.6.2 |
Hox-B6 (HOX2B) |
|
 |
T01733 |
 |
 |
|
| |
3.1.1.6.3 |
Hox-C6 (HOX3C) |
|
 |
T25653 |
 |
 |
|
| |
3.1.1.6.3.1 |
Hox-C6, isoform 1 (PRII) |
|
 |
T01744 |
|
| |
3.1.1.6.3.2 |
Hox-C6, isoform 2 (PRI) |
|
 |
T01743 |
| |
3.1.1.6.4 |
Hox-A7 (HOX1A) |
|
 |
T01278 |
 |
 |
|
| |
3.1.1.6.5 |
Hox-B7 (HOX2C) |
|
 |
T01735 |
 |
 |
|
| |
3.1.1.7 |
Subfamily: HOX8 |
GTAATTA |
|
| |
3.1.1.7.1 |
Hox-B8 (HOX2D) |
|
 |
T01737 |
 |
 |
|
| |
3.1.1.7.2 |
Hox-C8 (HOX3A) |
|
 |
T01749 |
 |
 |
|
| |
3.1.1.7.3 |
Hox-D8 (HOX4E) |
 |
 |
T |
 |
 F F |
|
| |
3.1.1.8 |
Subfamily: HOX9-13 |
TCGTAAA |
|
| |
3.1.1.8.1 |
Hox-A9 (HOX1G) |
|
 |
T14354 |
 |
 |
 |
| |
3.1.1.8.1.1 |
Hox-A9, isoform 1 |
|
 |
T01710 |
|
| |
3.1.1.8.1.2 |
Hox-A9, isoform 2 (T) |
|
 |
T04132 |
|
| |
3.1.1.8.2 |
Hox-B9 (HOX2E) |
|
 |
T01739 |
 |
 |
|
| |
3.1.1.8.3 |
Hox-C9 (HOX3B) |
|
 |
T03349 |
 |
 |
 |
| |
3.1.1.8.4 |
Hox-D9 (HOX4C) |
 |
 |
T01755 |
 |
 |
|
| |
3.1.1.8.5 |
Hox-A10 (HOX1H) |
 |
 |
T01715 |
 |
 |
|
| |
3.1.1.8.5.1 |
Hox-A10, isoform 1 |
|
 |
T09504 |
|
| |
3.1.1.8.5.2 |
Hox-A10, isoform 2 |
|
 |
T09505 |
|
| |
3.1.1.8.6 |
Hox-C10 (HOX3I) |
 |
 |
T03350 |
 |
 |
|
| |
3.1.1.8.7 |
Hox-D10 (HOX4D, HOX4E) |
|
 |
T01757 |
 |
 |
|
| |
3.1.1.8.8 |
Hox-A11 (HOX1I) |
 |
 |
T01717 |
 |
 |
|
| |
3.1.1.8.9 |
Hox-C11 (HOX3H) |
 |
 |
T03352 |
 |
 |
|
| |
3.1.1.8.10 |
Hox-D11 (HOX4F) |
 |
 |
T01760 |
 |
|
|
| |
3.1.1.8.11 |
Hox-C12 (HOX3F) |
 |
 |
T03353 |
 |
 |
|
| |
3.1.1.8.12 |
Hox-D12 (HOX4H) |
 |
 |
T01763 |
 |
 |
|
| |
3.1.1.8.13 |
Hox-A13 (HOX1J) |
 |
 |
T03337 |
 |
 F F |
 |
| |
3.1.1.8.14 |
Hox-B13 |
 |
 |
T03802 |
 |
 |
 |
| |
3.1.1.8.15 |
Hox-C13 (HOX3G) |
 |
 |
T03354 |
 |
 |
|
| |
3.1.1.8.16 |
Hox-D13 (HOX4I) |
 |
 |
T01765 |
 |
 |
|
| |
3.1.1.9 |
Subfamily: CDX (Caudal type homeobox) |
GGTAATAAA |
|
| |
3.1.1.9.1 |
CDX-1 |
 |
 |
T01999 |
 |
 F F |
|
| |
3.1.1.9.2 |
CDX-2 (CDX-3) |
|
 |
T02002 |
 |
 |
|
| |
3.1.1.9.3 |
CDX-4 |
|
 |
T03245 |
 |
 |
|
| |
3.1.1.10 |
Subfamily: EVX (Even-skipped homolog) |
CTAATTAG |
|
| |
3.1.1.10.1 |
EVX-1 |
 |
 |
T02023 |
 |
 |
|
| |
3.1.1.10.2 |
EVX-2 |
 |
 |
T16867 |
 |
 |
|
| |
3.1.1.11 |
Subfamily: GBX (Gastrulation brain homeobox) |
CTAATTAG |
|
| |
3.1.1.11.1 |
GBX-1 |
 |
 |
T16868 |
 |
 |
|
| |
3.1.1.11.2 |
GBX-2 |
 |
 |
T04020 |
 |
 |
|
| |
3.1.1.12 |
Subfamily: GSX |
TAATTA |
|
| |
3.1.1.12.1 |
GSX-1 (GSH-1) |
 |
 |
T05100 |
 |
 |
|
| |
3.1.1.12.2 |
GSX-2 (GSH-2) |
 |
 |
T17003 |
 |
 |
|
| |
3.1.1.13 |
Subfamily: MNX |
ACTAATTA |
|
| |
3.1.1.13.1 |
MNX1 (HB9, HLXB9) |
 |
 |
T03421 |
 |
 |
|
| |
3.1.1.14 |
Subfamily: MEOX |
TAATTA |
|
| |
3.1.1.14.1 |
MEOX-1 (MOX-1) |
 |
 |
T04047 |
 |
 |
|
| |
3.1.1.14.2 |
MEOX-2 (MOX-2) |
 |
 |
T04048 |
 |
 |
|
| |
3.1.1.15 |
Subfamily: PDX |
TAATTA |
|
| |
3.1.1.15.1 |
PDX-1 (IPF-1) |
 |
 |
T02057 |
 |
 |
 |
| |
3.1.2 |
Family: NK-related factors |
|
|
| |
3.1.2.1 |
Subfamily: BARHL |
AACCAATTA |
|
| |
3.1.2.1.1 |
BarH-L1 |
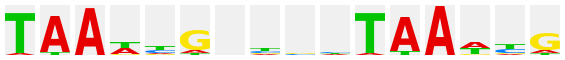 |
 |
T03920 |
 |
 |
 |
| |
3.1.2.1.2 |
BarH-L2 |
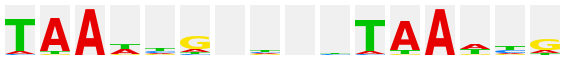 |
 |
T16779 |
 |
 |
|
| |
3.1.2.2 |
Subfamily: BARX |
TAATTA |
|
| |
3.1.2.2.1 |
BARX-1 |
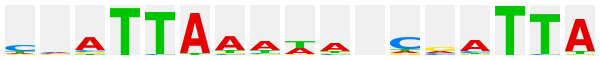 |
 |
T02403 |
 |
 |
|
| |
3.1.2.2.2 |
BARX-2 |
|
 |
T11228 |
 |
 |
|
| |
3.1.2.3 |
Subfamily: BSX |
TAATTA |
|
| |
3.1.2.3.1 |
BSX-1 (BSX) |
 |
 |
T16798 |
 |
 |
|
| |
3.1.2.3.1.1 |
BSX-1, isoform 1 (Bsxla) |
|
 |
T16797 |
|
| |
3.1.2.3.1.2 |
BSX-1, isoform 2 (Bsxlb) |
|
 |
T16800 |
|
| |
3.1.2.4 |
Subfamily: DBX |
TTAATTA |
|
| |
3.1.2.4.1 |
DBX-1 |
|
 |
T08090 |
 |
 |
|
| |
3.1.2.4.2 |
DBX-2 |
|
 |
T08092 |
 |
 |
|
| |
3.1.2.5 |
Subfamily: DLX |
TAATTA |
|
| |
3.1.2.5.1 |
DLX-1 |
 |
 |
T02006 |
 |
 |
 |
| |
3.1.2.5.2 |
DLX-2 |
 |
 |
T02009 |
 |
 |
|
| |
3.1.2.5.3 |
DLX-3 |
 |
 |
T02409 |
 |
 |
|
| |
3.1.2.5.4 |
DLX-4 |
 |
 |
T03993 |
 |
 |
|
| |
3.1.2.5.5 |
DLX-5 |
 |
 |
T03989 |
 |
 |
|
| |
3.1.2.5.5.1 |
DLX-5, isoform 1 (α) |
|
 |
T08471 |
|
| |
3.1.2.5.5.2 |
DLX-5, isoform 2 (β) |
|
 |
T17006 |
|
| |
3.1.2.5.5.3 |
DLX-5, isoform 3 (γ) |
|
 |
T17007 |
|
| |
3.1.2.5.6 |
DLX-6 |
 |
 |
T03990 |
 |
 F F |
|
| |
3.1.2.5.6.1 |
DLX-6, isoform 1 |
|
 |
T14717 |
|
| |
3.1.2.5.6.2 |
DLX-6, isoform 2 |
|
 |
T26086 |
|
| |
3.1.2.6 |
Subfamily: EMX |
CTAATTAG |
|
| |
3.1.2.6.1 |
EMX-1 |
 |
 |
T02011 |
 |
 |
|
| |
3.1.2.6.2 |
EMX-2 |
 |
 |
T02013 |
 |
 |
|
| |
3.1.2.7 |
Subfamily: EN (Engrailed-like factors) |
CTAATTAG |
|
| |
3.1.2.7.1 |
EN-1 |
 |
 |
T02016 |
 |
 F F |
|
| |
3.1.2.7.2 |
EN-2 |
 |
 |
T02020 |
 |
 |
|
| |
3.1.2.8 |
Subfamily: HHEX |
ATTAA |
|
| |
3.1.2.8.1 |
HHEX (PRH) |
|
 |
T03417 |
 |
 |
|
| |
3.1.2.9 |
Subfamily: HLX |
TAATTAATTA |
|
| |
3.1.2.9.1 |
HLX-1 (HB24) |
|
 |
T02053 |
 |
 |
|
| |
3.1.2.10 |
Subfamily: LBX |
TTAATTAA |
|
| |
3.1.2.10.1 |
LBX-1 |
|
 |
T04315 |
 |
 |
|
| |
3.1.2.10.2 |
LBX-2 |
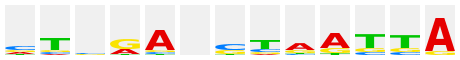 |
 |
T16903 |
 |
 |
|
| |
3.1.2.11 |
Subfamily: MSX |
ACCAATTA |
|
| |
3.1.2.11.1 |
MSX-1 (HOX7) |
 |
 |
T02072 |
 |
 |
 |
| |
3.1.2.11.2 |
MSX-2 (HOX8) |
 |
 |
T02076 |
 |
 |
|
| |
3.1.2.12 |
Subfamily: NANOG |
CCATTT |
|
| |
3.1.2.12.1 |
NANOG |
|
 |
|
 |
 |
 |
| |
3.1.2.12.1.1 |
NANOG, isoform 1 |
|
 |
|
|
| |
3.1.2.12.1.2 |
NANOG, isoform 2 |
|
 |
|
|
| |
3.1.2.13 |
Subfamily: NK-1 |
CTAATTAG |
|
| |
3.1.2.13.1 |
NKX-1.1 (NKX1-1, SAX-2) |
|
 |
T16916 |
 |
 |
|
| |
3.1.2.13.2 |
NKX-1.2 (NKX1-2, SAX-1) |
|
 |
T16801 |
 |
 |
|
| |
3.1.2.14 |
Subfamily: NK-2.1 |
CCACTTGA |
|
| |
3.1.2.14.1 |
NKX-2.1 (NKX2-1, TTF-1) |
|
 |
T00859 |
 |
 |
 |
| |
3.1.2.14.2 |
NKX-2.4 (NKX2-4, NK-2HD) |
|
 |
T16918 |
 |
 |
|
| |
3.1.2.15 |
Subfamily: NK-2.2 |
CCACTTGA |
|
| |
3.1.2.15.1 |
NKX-2.2 (NKX2-2, NKX2B) |
|
 |
T02384 |
 |
 |
|
| |
3.1.2.15.2 |
NKX-2.8 (NKX2-8, NKX2-9, NKX2G, NKX2H) |
 |
 |
T04338 |
 |
 |
|
| |
3.1.2.16 |
Subfamily: NK-3 |
AAGTACTT |
|
| |
3.1.2.16.1 |
NKX-3.1 (NKX3-1, NKX3A) |
 |
 |
T04256 |
 |
 |
 |
| |
3.1.2.16.2 |
NKX-3.2 (NKX3-2, NKX3B) |
 |
 |
T02667 |
 |
 F F |
|
| |
3.1.2.17 |
Subfamily: NK-4 |
CCACTTAA |
|
| |
3.1.2.17.1 |
NKX-2.3 (NKX2-3, NKX2C, NKX4-3) |
 |
 |
T04325 |
 |
 |
|
| |
3.1.2.17.2 |
NKX-2.5 (NKX2-5, NKX2E, NKX4-1, CSX) |
|
 |
T01675 |
 |
 |
 |
| |
3.1.2.17.3 |
NKX-2.6 (NKX2-6, NKX2F, NKX4-2, CSX2) |
|
 |
T04342 |
 |
 |
|
| |
3.1.2.18 |
Subfamily: NK-5/HMX |
AGCAATTAA |
|
| |
3.1.2.18.1 |
HMX-1 (H6, NKX5-3, NKX-5.3) |
 |
 |
T04444 |
 |
 |
|
| |
3.1.2.18.2 |
HMX-2 (H6L, NKX5-2, NKX-5.2) |
 |
 |
T22946 |
 |
 |
|
| |
3.1.2.18.3 |
HMX-3 (NKX5-1, NKX-5.1) |
 |
 |
T04446 |
 |
 |
|
| |
3.1.2.19 |
Subfamily: NK-6 |
AATAATTA |
|
| |
3.1.2.19.1 |
NKX-6.1 (NKX6-1, NKX6A) |
 |
 |
T04269 |
 |
 |
|
| |
3.1.2.19.2 |
NKX-6.2 (NKX6-2, NKX6B) |
 |
 |
T02050 |
 |
 |
|
| |
3.1.2.19.3 |
NKX-6.3 (NKX6-3) |
|
 |
T16930 |
 |
 |
|
| |
3.1.2.20 |
Subfamily: NOTO |
|
|
| |
3.1.2.20.1 |
NOTO |
 |
 |
T19770 |
 |
 |
|
| |
3.1.2.21 |
Subfamily: TLX |
AATTAATA |
|
| |
3.1.2.21.1 |
TLX-1 (HOX11, TCL-3) |
|
 |
T02055 |
 |
 |
|
| |
3.1.2.21.2 |
TLX-2 (HOX11L1, NCX) |
|
 |
T04368 |
 |
 |
 |
| |
3.1.2.21.3 |
TLX-3 (HOX11L2) |
|
 |
T04314 |
 |
 |
|
| |
3.1.2.22 |
Subfamily: VAX |
TAATTA |
|
| |
3.1.2.22.1 |
VAX-1 |
 |
 |
T03274 |
 |
 |
|
| |
3.1.2.22.1.1 |
VAX-1, isoform 1 |
|
 |
T14527 |
|
| |
3.1.2.22.1.2 |
VAX-1, isoform 2 |
|
 |
T26350 |
|
| |
3.1.2.22.2 |
VAX-2 |
 |
 |
T03276 |
 |
 |
|
| |
3.1.2.23 |
Subfamily: VENTX |
|
|
| |
3.1.3 |
Family: Paired-related HD factors |
|
|
| |
3.1.3.1 |
Subfamily: ALX |
TTAATTAA |
|
| |
3.1.3.1.1 |
ALX-1 (CART-1) |
 |
 |
T03999 |
 |
 |
|
| |
3.1.3.1.2 |
ALX-3 |
 |
 |
T03343 |
 |
 |
|
| |
3.1.3.1.3 |
ALX-4 |
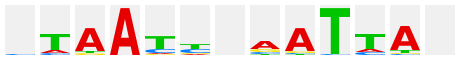 |
 |
T02967 |
 |
 |
|
| |
3.1.3.2 |
Subfamily: ARGFX |
|
|
| |
3.1.3.3 |
Subfamily: ARX |
TTAATTAA |
|
| |
3.1.3.3.1 |
ARX |
|
 |
T16770 |
 |
 |
|
| |
3.1.3.4 |
Subfamily: DMBX |
GGATTA |
|
| |
3.1.3.4.1 |
DMBX (MBX, OTX-3, PAXB) |
 |
 |
T06253 |
 |
 |
|
| |
3.1.3.4.1.1 |
DMBX, isoform 1 |
|
 |
T11177 |
|
| |
3.1.3.4.1.2 |
DMBX, isoform 2 |
|
 |
T26087 |
|
| |
3.1.3.5 |
Subfamily: DPRX |
|
|
| |
3.1.3.6 |
Subfamily: DRGX |
|
|
| |
3.1.3.6.1 |
DRGX (PRRXL1, Drg11) |
 |
 |
T19744 |
 |
 |
|
| |
3.1.3.6.1.1 |
DRGX, isoform 1 |
|
 |
T25980 |
|
| |
3.1.3.6.1.2 |
DRGX, isoform 2 (Prrxl1-B) |
|
 |
T19743 |
|
| |
3.1.3.7 |
Subfamily: DUX |
TCTAATTGAGAATTAC |
|
| |
3.1.3.8 |
Subfamily: ESX |
TTAATTAA |
|
| |
3.1.3.9 |
Subfamily: GSC |
GGATTA |
|
| |
3.1.3.9.1 |
GSC |
 |
 |
T02047 |
 |
 |
|
| |
3.1.3.9.2 |
GSC-2 |
 |
 |
T04039 |
 |
 |
|
| |
3.1.3.10 |
Subfamily: HESX |
TAATTGAATTA |
|
| |
3.1.3.10.1 |
HESX-1 |
 |
 |
T02785 |
 |
 |
 |
| |
3.1.3.11 |
Subfamily: HOPX |
|
|
| |
3.1.3.11.1 |
HOPX-1 |
|
 |
T |
 |
 |
 |
| |
3.1.3.12 |
Subfamily: ISX |
CTAATTAG |
|
| |
3.1.3.12.1 |
ISX |
 |
 |
T17019 |
 |
 |
|
| |
3.1.3.12.1.1 |
ISX, isoform 1 |
|
 |
T17018 |
|
| |
3.1.3.12.1.2 |
ISX, isoform 2 |
|
 |
T26163 |
|
| |
3.1.3.13 |
Subfamily: LEUTX |
|
|
| |
3.1.3.14 |
Subfamily: MIX |
|
|
| |
3.1.3.14.1 |
MIXL1 |
 |
 |
T04800 |
 |
 |
|
| |
3.1.3.15 |
Subfamily: NOBOX |
CTAATTAG |
|
| |
3.1.3.16 |
Subfamily: OTP |
TTAATTAA |
|
| |
3.1.3.16.1 |
OTP |
|
 |
T16920 |
 |
 |
|
| |
3.1.3.17 |
Subfamily: OTX |
GGGATTAA |
|
| |
3.1.3.17.1 |
OTX-1 |
 |
 |
T02080 |
 |
 |
|
| |
3.1.3.17.2 |
OTX-2 |
 |
 |
T02083 |
 |
 |
 |
| |
3.1.3.17.3 |
CRX (CORD-2) |
|
 |
T03461 |
 |
 |
|
| |
3.1.3.18 |
Subfamily: PHOX |
ATAATTAA |
|
| |
3.1.3.18.1 |
PHOX-2A (ARIX, PMX2A) |
 |
 |
T02090 |
 |
 |
|
| |
3.1.3.18.2 |
PHOX-2B (PMX2B) |
 |
 |
T03976 |
 |
 |
|
| |
3.1.3.19 |
Subfamily: PITX |
GGGATTAA |
|
| |
3.1.3.19.1 |
PITX-1 (BFT, PTX1) |
 |
 |
T02415 |
 |
 |
|
| |
3.1.3.19.2 |
PITX-2 (ARP, RGS, RIEG1, Ptx2) |
|
 |
T09186 |
 |
 |
 |
| |
3.1.3.19.2.1 |
Ptx2A |
|
 |
T02410 |
|
| |
3.1.3.19.2.2 |
Ptx2B |
|
 |
T02412 |
|
| |
3.1.3.19.2.3 |
Ptx2C |
|
 |
T17005 |
|
| |
3.1.3.19.2.4 |
Ptx2Cα |
|
 |
T25959 |
|
| |
3.1.3.19.2.5 |
Ptx2Cβ |
|
 |
T25960 |
|
| |
3.1.3.19.3 |
PITX-3 (PTX3) |
 |
 |
T02666 |
 |
 |
|
| |
3.1.3.20 |
Subfamily: PROP |
TTAATTAA |
|
| |
3.1.3.20.1 |
PROP-1 |
 |
 |
T |
 |
 |
|
| |
3.1.3.21 |
Subfamily: PRRX |
GCTAATTAGC |
|
| |
3.1.3.21.1 |
PRRX-1 (PHOX1, PMX1) |
 |
 |
T02966 |
 |
 |
|
| |
3.1.3.21.1.1 |
PRRX-1, isoform 1 (K2-a) |
|
 |
T02060 |
|
| |
3.1.3.21.1.2 |
PRRX-1, isoform 2 (K2-b) |
|
 |
T02061 |
|
| |
3.1.3.21.2 |
PRRX-2 (PMX2, PRX2) |
 |
 |
T01483 |
 |
 |
|
| |
3.1.3.22 |
Subfamily: RAX |
CTAATTAG |
|
| |
3.1.3.22.1 |
RAX (RX) |
 |
 |
T03502 |
 |
 |
|
| |
3.1.3.23 |
Subfamily: RHOX |
CTAATTAG |
|
| |
3.1.3.24 |
Subfamily: SEBOX |
|
|
| |
3.1.3.24.1 |
SEBOX (OG-9) |
|
 |
T03309 |
 |
 |
|
| |
3.1.3.25 |
Subfamily: SHOX |
TTAATTAA |
|
| |
3.1.3.25.2 |
SHOX-2 (OG-12X, SHOT) |
 |
 |
T14734 |
 |
 |
|
| |
3.1.3.25.2.1 |
SHOX-2, isoform 1 (OG12A) |
|
 |
T03310 |
|
| |
3.1.3.25.2.2 |
SHOX-2, isoform 2 (OG12B) |
|
 |
T26289 |
|
| |
3.1.3.26 |
Subfamily: TPRX |
|
|
| |
3.1.3.27 |
Subfamily: UNCX |
TTAATTAA |
|
| |
3.1.3.27.1 |
UNCX (Uncx4.1) |
 |
 |
T03307 |
 |
 |
|
| |
3.1.3.28 |
Subfamily: VSX |
TTAATTAA |
|
| |
3.1.3.28.1 |
VSX-1 (RINX) |
 |
 |
T16785 |
 |
 |
|
| |
3.1.3.28.2 |
VSX-2 (HOX10) |
 |
 |
T02970 |
 |
 |
|
| |
3.1.4 |
Family: TALE-type homeo domain factors |
|
|
| |
3.1.4.1 |
Subfamily: IRX (Iroquois) |
TACATGTA |
|
| |
3.1.4.1.1 |
IRX-1 |
|
 |
T04281 |
 |
 |
|
| |
3.1.4.1.2 |
IRX-2 |
 |
 |
T14754 |
 |
 |
|
| |
3.1.4.1.3 |
IRX-3 |
 |
 |
T02439 |
 |
 |
|
| |
3.1.4.1.3.1 |
IRX-3, isoform 1 |
|
 |
T11255 |
|
| |
3.1.4.1.3.2 |
IRX-3, isoform 2 |
|
 |
T26162 |
|
| |
3.1.4.1.4 |
IRX-4 |
|
 |
T04276 |
 |
 |
|
| |
3.1.4.1.5 |
IRX-5 |
 |
 |
T04283 |
 |
 |
|
| |
3.1.4.1.6 |
IRX-6 |
|
 |
T08164 |
 |
 |
|
| |
3.1.4.2 |
Subfamily: MEIS |
AGCTGTCAA |
|
| |
3.1.4.2.1 |
Meis1 |
 |
 |
T09993 |
 |
 |
|
| |
3.1.4.2.1.1 |
Meis1, isoform 1 (Meis1A) |
|
 |
T03388 |
|
| |
3.1.4.2.1.2 |
Meis1, isoform 2 (Meis1B) |
|
 |
T03389 |
|
| |
3.1.4.2.1.3 |
Meis1, isoform 3 |
|
 |
T26190 |
|
| |
3.1.4.2.2 |
Meis2 |
 |
 |
T11160 |
 |
 |
 |
| |
3.1.4.2.2.1 |
Meis2A |
|
 |
T03405 |
|
| |
3.1.4.2.2.2 |
Meis2B |
|
 |
T03407 |
|
| |
3.1.4.2.2.3 |
Meis2C |
|
 |
T03408 |
|
| |
3.1.4.2.2.4 |
Meis2D |
|
 |
T03409 |
|
| |
3.1.4.2.3 |
Meis3 |
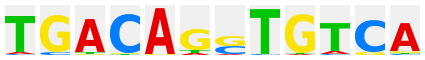 |
 |
T03406 |
 |
 |
|
| |
3.1.4.2.3.1 |
Meis3, isoform 1 |
|
 |
T11188 |
|
| |
3.1.4.2.3.2 |
Meis3, isoform 2 |
|
 |
T25908 |
|
| |
3.1.4.3 |
Subfamily: MKX (Mohawk) |
|
|
| |
3.1.4.3.1 |
MKX (IRX-L1) |
|
 |
T |
 |
 |
|
| |
3.1.4.4 |
Subfamily: PBX |
CATCAAT |
|
| |
3.1.4.4.1 |
PBX-1 |
|
 |
T08131 |
 |
 |
 |
| |
3.1.4.4.1.1 |
PBX-1a |
|
 |
T03451 |
|
| |
3.1.4.4.1.2 |
PBX-1b |
|
 |
T02088 |
|
| |
3.1.4.4.2 |
PBX-2 |
|
 |
T04127 |
 |
 |
|
| |
3.1.4.4.3 |
PBX-3 |
|
 |
T14296 |
 |
 |
|
| |
3.1.4.4.3.1 |
PBX-3a |
|
 |
T04125 |
|
| |
3.1.4.4.3.2 |
PBX-3b |
|
 |
T04126 |
|
| |
3.1.4.4.4 |
PBX-4 |
|
 |
T19780 |
 |
 |
|
| |
3.1.4.4.4.1 |
PBX-4, isoform 1 |
|
 |
T19779 |
|
| |
3.1.4.4.4.2 |
PBX-4, isoform 2 |
|
 |
T19781 |
|
| |
3.1.4.5 |
Subfamily: PKNOX |
ACCTGTCAAT |
|
| |
3.1.4.5.1 |
PKNOX-1 (PREP-1) |
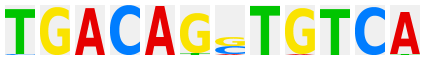 |
 |
T04121 |
 |
 |
 |
| |
3.1.4.5.2 |
PKNOX-2 (PREP-2) |
 |
 |
T16934 |
 |
 |
|
| |
3.1.4.6 |
Subfamily: TGIF |
AGCTGTCAAT |
|
| |
3.1.4.6.1 |
TGIF-1 |
 |
 |
T04077 |
 |
 |
 |
| |
3.1.4.6.1.1 |
TGIF-1, isoform 1 |
|
 |
T10218 |
|
| |
3.1.4.6.1.2 |
TGIF-1, isoform 2 |
|
 |
T17016 |
|
| |
3.1.4.6.2 |
TGIF-2 |
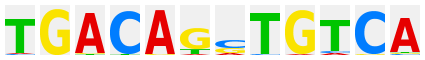 |
 |
T16763 |
 |
 |
|
| |
3.1.4.6.3 |
TGIF-2LX |
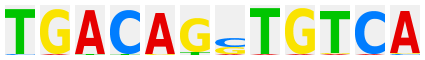 |
 |
T |
 |
 |
 |
| |
3.1.5 |
Family: HD-LIM factors |
AATTAA; |
|
| |
3.1.5.1 |
Subfamily: ISL |
ATCAATTAA |
|
| |
3.1.5.1.1 |
ISL-1 (Islet-1) |
|
 |
T09161 |
 |
 |
|
| |
3.1.5.1.1.1 |
ISL-1, isoform 1 (α) |
|
 |
T06054 |
|
| |
3.1.5.1.1.2 |
ISL-1, isoform 2 (β) |
|
 |
T06055 |
|
| |
3.1.5.1.2 |
ISL-2 (Islet-2) |
 |
 |
T16901 |
 |
 |
|
| |
3.1.5.2 |
Subfamily: Lhx-1-like factors |
TTAATTAA |
|
| |
3.1.5.2.1 |
Lhx1 (Lim-1) |
|
 |
T01961 |
 |
 |
|
| |
3.1.5.2.2 |
Lhx5 |
|
 |
T04181 |
 |
 |
|
| |
3.1.5.3 |
Subfamily: Lhx-2-like factors |
TAATTA |
|
| |
3.1.5.3.1 |
Lhx2 (LH-2) |
 |
 |
T01969 |
 |
 |
|
| |
3.1.5.3.2 |
Lhx9 |
 |
 |
T11357 |
 |
 |
 |
| |
3.1.5.3.2.1 |
Lhx9, isoform 1 β |
|
 |
T04192 |
|
| |
3.1.5.3.2.2 |
Lhx9, isoform 2 α |
|
 |
T04195 |
|
| |
3.1.5.3.2.3 |
Lhx9, isoform 3 |
|
 |
T25470 |
|
| |
3.1.5.3.2.4 |
Lhx9, isoform 4 |
|
 |
T26174 |
|
| |
3.1.5.4 |
Subfamily: Lhx-3-like factors |
TAATTA |
|
| |
3.1.5.4.1 |
Lhx3 |
|
 |
T10879 |
 |
 |
|
| |
3.1.5.4.1.1 |
Lhx3, isoform A |
|
 |
T01964 |
|
| |
3.1.5.4.1.2 |
Lhx3, isoform B |
|
 |
T04840 |
|
| |
3.1.5.4.2 |
Lhx4 |
 |
 |
T03542 |
 |
 |
|
| |
3.1.5.5 |
Subfamily: Lhx-6-like factors |
CTAATTAG |
|
| |
3.1.5.5.1 |
Lhx6 (Lhx6.1) |
 |
 |
T05162 |
 |
 |
|
| |
3.1.5.5.1.1 |
Lhx6, isoform 1 (Lhx6.1a) |
|
 |
T04187 |
|
| |
3.1.5.5.1.2 |
Lhx6, isoform 2 (Lhx6.1b) |
|
 |
T04188 |
|
| |
3.1.5.5.2 |
Lhx8 |
 |
 |
T11354 |
 |
 |
|
| |
3.1.5.6 |
Subfamily: Lmx |
TTAATTAA |
|
| |
3.1.5.6.1 |
Lmx-1α (LMX-1.1, LMX1A) |
 |
 |
T04300 |
 |
 |
|
| |
3.1.5.6.2 |
Lmx-1β (LMX-1.2, LMX1B) |
 |
 |
T04303 |
 |
 |
|
| |
3.1.6 |
Family: HD-SINE factors |
GGTATCA |
|
| |
3.1.6.1 |
Subfamily: SIX1-like factors |
GGGTATCA |
|
| |
3.1.6.1.1 |
SIX1 |
|
 |
T03262 |
 |
|
|
| |
3.1.6.1.2 |
SIX2 |
|
 |
T03261 |
 |
 |
|
| |
3.1.6.2 |
Subfamily: SIX3-like factors |
GGGTATCA |
|
| |
3.1.6.2.1 |
SIX3 |
|
 |
T09227 |
 |
 |
|
| |
3.1.6.2.1.1 |
SIX3A (α) |
|
 |
T03263 |
|
| |
3.1.6.2.1.2 |
SIX3B (β) |
|
 |
T03270 |
|
| |
3.1.6.2.2 |
SIX6 |
|
 |
T03272 |
 |
 |
|
| |
3.1.6.3 |
Subfamily: SIX4-like factors |
GGTGTCA |
|
| |
3.1.6.3.1 |
SIX4 |
|
 |
T08392 |
 |
 |
|
| |
3.1.6.3.1.1 |
SIX4, isoform 1 (SM) |
|
 |
T03264 |
|
| |
3.1.6.3.1.2 |
SIX4, isoform 2 (M18) |
|
 |
T03265 |
|
| |
3.1.6.3.1.3 |
SIX4, isoform 3 (M8) |
|
 |
T03266 |
|
| |
3.1.6.3.2 |
SIX5 |
|
 |
T03271 |
 |
 |
|
| |
3.1.7 |
Family: HD-PROS factors |
|
|
| |
3.1.7.0.1 |
PROX-1 |
 |
 |
T |
 |
 |
 |
| |
3.1.7.0.2 |
PROX-2 |
|
 |
T |
 |
 |
|
| |
3.1.8 |
Family: HD-ZF factors |
|
|
| |
3.1.8.1 |
Subfamily: ADNP |
|
|
| |
3.1.8.1.1 |
ADNP1 = 2.3.4.0.77 |
|
 |
T21522 |
 |
 |
|
| |
3.1.8.1.2 |
ADNP2 (ZNF508) = 2.3.4.0.78 |
|
 |
T19732 |
 |
 |
|
| |
3.1.8.2 |
Subfamily: TSHZ |
|
|
| |
3.1.8.2.1 |
TSHZ-1 |
|
 |
T19791 |
 |
 |
|
| |
3.1.8.2.2 |
TSHZ-2 |
|
 |
T19795 |
 |
 F F |
|
| |
3.1.8.2.3 |
TSHZ-3 |
|
 |
T19799 |
 |
 |
|
| |
3.1.8.3 |
Subfamily: ZEB |
CACCTG |
|
| |
3.1.8.3.1 |
ZEB1 (TCF-8) |
|
 |
T03915 |
 |
 |
 |
| |
3.1.8.3.2 |
ZEB2 (SMADIP1) |
|
 |
T04864 |
 |
 |
 |
| |
3.1.8.4 |
Subfamily: ZFHX |
|
|
| |
3.1.8.4.1 |
ZFHX2 (ZFH-2) |
|
 |
T |
 |
 |
|
| |
3.1.8.4.2 |
ZFHX3 (ZFH-3, ATBF1) |
|
 |
T03881 |
 |
 |
|
| |
3.1.8.4.3 |
ZFHX4 (ZFH-4) |
|
 |
T21506 |
 |
 |
|
| |
3.1.8.5 |
Subfamily: ZHX |
ATCGTTTT |
|
| |
3.1.8.5.1 |
ZHX1 |
|
 |
T04356 |
 |
 |
 |
| |
3.1.8.5.2 |
ZHX2 (AFP) |
|
 |
T19824 |
 |
 |
 |
| |
3.1.8.5.3 |
ZHX3 (TIX1) |
|
 |
T19826 |
 |
 |
 |
| |
3.1.8.5.4 |
HOMEZ |
 |
 |
T16893 |
 |
 |
 |
| |
3.1.8.5.4.1 |
HOMEZ, isoform 1 |
|
 |
T16892 |
|
| |
3.1.8.5.4.2 |
HOMEZ, isoform 2 |
|
 |
T16896 |
|
| |
3.1.9 |
Family: HD-CUT factors |
|
|
| |
3.1.9.1 |
Subfamily: ONECUT |
AAATCAAT |
|
| |
3.1.9.1.1 |
ONECUT-1 (HNF6, HNF-6α, OC-1) |
 |
 |
T03296 |
 |
 |
 |
| |
3.1.9.1.2 |
ONECUT-2 (HNF-6β, OC-2) |
 |
 |
T16615 |
 |
 F F |
|
| |
3.1.9.1.3 |
ONECUT-3 (OC-3) |
 |
 |
T19774 |
 |
 |
|
| |
3.1.9.2 |
Subfamily: CUX |
TGATCA |
|
| |
3.1.9.2.1 |
Cux-1 (CDP, Cut-L1) |
 |
 |
T09991 |
 |
 F F |
|
| |
3.1.9.2.1.1 |
Cux-1, isoform 1 (CASP) |
|
 |
T17015 |
|
| |
3.1.9.2.1.2 |
Cux-1, isoform 2 |
|
 |
T17013 |
|
| |
3.1.9.2.1.3 |
Cux-1, isoform 3 |
|
 |
T26079 |
|
| |
3.1.9.2.1.4 |
Cux-1, isoform 4 (CASP) |
|
 |
T02042 |
|
| |
3.1.9.2.1.5 |
Cux-1, isoform 5 |
|
 |
T17012 |
|
| |
3.1.9.2.1.6 |
Cux-1, isoform 6 |
|
 |
T17014 |
|
| |
3.1.9.2.2 |
Cux-2 (Cut-L2) |
 |
 |
T04456 |
 |
 F F |
 |
| |
3.1.9.3 |
Subfamily: SATB |
TAATGA |
|
| |
3.1.9.3.1 |
SATB-1 |
|
 |
T17580 |
 |
 |
|
| |
3.1.9.3.2 |
SATB-2 |
|
 |
T |
 |
 |
|
| |
3.1.9.3.2.1 |
SATB-2, isoform 1 |
|
 |
T |
|
| |
3.1.9.3.2.2 |
SATB-2, isoform 2 |
|
 |
T |
|
| |
3.1.10 |
Family: POU domain factors |
|
|
| |
3.1.10.1 |
Subfamily: POU1 (Pit-1-like factors) |
TAATTAATTA |
|
| |
3.1.10.1.1 |
POU1F1 (Pit-1, GHF-1) |
 |
 |
T01172 |
 |
 |
|
| |
3.1.10.1.1.1 |
POU1F1, isoform 1 (Pit-1) |
|
 |
T09174 |
|
| |
3.1.10.1.1.2 |
POU1F1, isoform 2 (Pit-1T) |
|
 |
T26243 |
|
| |
3.1.10.1.1.3 |
POU1F1, isoform 3 (Pit-1a) |
|
 |
T17004 |
|
| |
3.1.10.2 |
Subfamily: POU2 (Oct-1/2-like factors) |
TATGCAAAT |
|
| |
3.1.10.2.1 |
POU2F1 (Oct-1, OTF-1) |
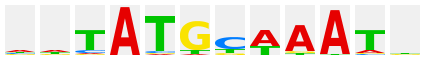 |
 |
T01192 |
 |
 |
 |
| |
3.1.10.2.1.1 |
POU2F1, isoform 1 (Oct-1A) |
|
 |
T00644 |
|
| |
3.1.10.2.1.2 |
POU2F1, isoform 2 (Oct-1B) |
|
 |
T01862 |
|
| |
3.1.10.2.1.3 |
POU2F1, isoform 3 (Oct-1C) |
|
 |
T01863 |
|
| |
3.1.10.2.1.4 |
POU2F1, isoform 4 |
|
 |
T26248 |
|
| |
3.1.10.2.1.5 |
POU2F1, isoform 5 (Oct-1R) |
|
 |
T26249 |
|
| |
3.1.10.2.1.6 |
POU2F1, isoform 6 |
|
 |
T26250 |
|
| |
3.1.10.2.1.7 |
POU2F1, isoform 7 |
|
 |
T26251 |
|
| |
3.1.10.2.1.8 |
POU2F1, isoform 8 |
|
 |
T26252 |
|
| |
3.1.10.2.1.9 |
POU2F1, isoform 9 |
|
 |
T26253 |
|
| |
3.1.10.2.1.10 |
POU2F1, isoform 10 (Oct-1Z) |
|
 |
T26387 |
|
| |
3.1.10.2.1.11 |
POU2F1, isoform 11 |
|
 |
T26388 |
|
| |
3.1.10.2.1.12 |
POU2F1, isoform 12 |
|
 |
T26389 |
|
| |
3.1.10.2.2 |
POU2F2 (Oct-2, OTF-2) |
 |
 |
T13860 |
 |
 |
|
| |
3.1.10.2.2.1 |
POU2F2, isoform 1 (OCT2.1) |
|
 |
T01864 |
|
| |
3.1.10.2.2.2 |
POU2F2, isoform 2 (OCT2.2) |
|
 |
T00648 |
|
| |
3.1.10.2.2.3 |
POU2F2, isoform 3 (OCT2.3) |
|
 |
T01865 |
|
| |
3.1.10.2.2.4 |
POU2F2, isoform 4 (OCT2.4) |
|
 |
T01866 |
|
| |
3.1.10.2.2.5 |
POU2F2, isoform 5 (OCT2.5) |
|
 |
T00964 |
|
| |
3.1.10.2.2.6 |
POU2F2, isoform 6 (OCT2.6) |
|
 |
T01867 |
|
| |
3.1.10.2.2.7 |
POU2F2, isoform 7 (OCT2.7) |
|
 |
T26254 |
|
| |
3.1.10.2.3 |
POU2F3 (Oct-11, OTF-11, Skn-1) |
 |
 |
T01874 |
 |
 |
|
| |
3.1.10.3 |
Subfamily: POU3 (Oct-6-like factors) |
TAATTAATTA |
|
| |
3.1.10.3.1 |
POU3F1 (Oct-6, OTF-6, SCIP) |
 |
 |
T18500 |
 |
 |
 |
| |
3.1.10.3.2 |
POU3F2 (Oct-7, OTF-7, Brn-2) |
 |
 |
T01873 |
 |
 |
|
| |
3.1.10.3.3 |
POU3F3 (Oct-8, OTF-8, Brn-1) |
 |
 |
T00658 |
 |
 |
|
| |
3.1.10.3.4 |
POU3F4 (Oct-9, OTF-9, Brn-4) |
 |
 |
T00659 |
 |
 |
|
| |
3.1.10.4 |
Subfamily: POU4 (Brn-3-like factors) |
ATTAATGA |
|
| |
3.1.10.4.1 |
POU4F1 (Brn-3A, Oct-T1) |
 |
 |
T06590 |
 |
 |
|
| |
3.1.10.4.2 |
POU4F2 (Brn-3B) |
 |
 |
T01881 |
 |
 |
|
| |
3.1.10.4.3 |
POU4F3 (Brn-3C) |
 |
 |
T04462 |
 |
 |
|
| |
3.1.10.5 |
Subfamily: POU5 (Oct-3/4-like factors) |
|
|
| |
3.1.10.5.1 |
POU5F1 (Oct-3, Oct-4, OTF-3) |
|
 |
T00546 |
 |
 |
 |
| |
3.1.10.5.3 |
POU5F2 (SPRM-1) |
|
 |
T19723 |
 |
 |
|
| |
3.1.10.6 |
Subfamily: POU6 (Brn-5-like factors) |
TAATGAG |
|
| |
3.1.10.6.1 |
POU6F1 (Brn-5) |
|
 |
T11287 |
 |
 |
 |
| |
3.1.10.6.1.1 |
POU6F1, isoform C2 |
|
 |
T01888 |
|
| |
3.1.10.6.1.2 |
POU6F1, isoform C1 |
|
 |
T01889 |
|
| |
3.1.10.6.1.3 |
POU6F1, isoform C7 |
|
 |
T01890 |
|
| |
3.1.10.6.2 |
POU6F2 (Brn-5) |
 |
 |
T19728 |
 |
 |
|
| |
3.1.10.7 |
Subfamily: HNF1-like factors |
GTTAATGATTAAC |
|
| |
3.1.10.7.1 |
HNF-1α (HNF-1A, LF-B1) |
 |
 |
T01211 |
 |
 |
|
| |
3.1.10.7.2 |
HNF-1β (HNF-1B, vHNF1, LF-B3) |
 |
 |
T00890 |
 |
 |
 |
| |
3.1.10.7.2.1 |
HNF-1β, isoform 1 |
|
 |
T09049 |
|
| |
3.1.10.7.2.2 |
HNF-1β, isoform 2 |
|
 |
T10376 |
|
| |
3.1.10.7.2.3 |
HNF-1β, isoform 3 |
|
 |
T17010 |
|
| |
3.1.10.7.2.4 |
HNF-1β, isoform 4 |
|
 |
T17011 |
|
| |
3.1.10.7.3 |
HMBOX1 (HNF-1LA) |
 |
 |
T16882 |
 |
 |
|
| |
3.1.10.7.3.1 |
HMBOX1, isoform 1 |
|
 |
T16881 |
|
| |
3.1.10.7.3.2 |
HMBOX1, isoform 2 |
|
 |
T16885 |
|
| |
3.1.10.7.3.3 |
HMBOX1, isoform 3 |
|
 |
T16887 |
|
| |
3.1.10.7.3.4 |
HMBOX1, isoform 4 |
|
 |
T16888 |
|
| |
3.1.10.8 |
Subfamily: HDX-like factors |
GAAATCA |
|
| |
3.1.10.8.1 |
HDX |
|
 |
T16877 |
 |
 |
|
| |
3.1.10.8.1.1 |
HDX, isoform 1 |
|
 |
T16876 |
|
| |
3.1.10.8.1.2 |
HDX, isoform 2 |
|
 |
T16878 |
|
| |
3.1.11 |
Family: PHTF factors |
|
|
| |
3.1.11.0.1 |
PHTF1 |
|
 |
T27847 |
 |
 |
|
| |
3.1.11.0.2 |
PHTF2 |
|
 |
T27848 |
 |
 |
|
| |
3.2 |
Class: Paired box factors |
 |
 |
| |
3.2.1 |
Family: Paired plus homeo domain |
CTAATTAG |
|
| |
3.2.1.1 |
Subfamily: PAX-3/7 |
CTAATTAG |
|
| |
3.2.1.1.1 |
Pax-3 (HuP2) |
 |
 |
T00680 |
 |
 |
 |
| |
3.2.1.1.2 |
Pax-7 (HuP1) |
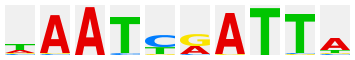 |
 |
T02960 |
 |
 |
|
| |
3.2.1.2 |
Subfamily: PAX-4/6 |
CTAATTAG |
|
| |
3.2.1.2.1 |
Pax-4 |
 |
 |
T11179 |
 |
 |
|
| |
3.2.1.2.1.1 |
Pax-4, isoform 1 |
|
 |
T02983 |
|
| |
3.2.1.2.1.2 |
Pax-4, isoform 2 |
|
 |
T26236 |
|
| |
3.2.1.2.1.3 |
Pax-4, isoform 3 |
|
 |
T03195 |
|
| |
3.2.1.2.2 |
Pax-6 (AN2) |
 |
 |
T00681 |
 |
 |
 |
| |
3.2.1.2.2.1 |
Pax-6, isoform 1 |
|
 |
T09150 |
|
| |
3.2.1.2.2.2 |
Pax-6, isoform 3 (p32) |
|
 |
|
|
| |
3.2.1.2.2.3 |
Pax-6-5a, isoform 5a |
|
 |
T09981 |
|
| |
3.2.2 |
Family: Paired domain only |
|
|
| |
3.2.2.1 |
Subfamily: PAX-1/9 (no homeo remnant) |
|
|
| |
3.2.2.1.1 |
Pax-1 (HuP48) |
 |
 |
T00677 |
 |
 |
|
| |
3.2.2.1.2 |
Pax-9 |
 |
 |
T02985 |
 |
 |
|
| |
3.2.2.2 |
Subfamily: PAX-2-like factors (partial homeobox) |
GTCATGCGTGA |
|
| |
3.2.2.2.1 |
Pax-2 |
 |
 |
T16059 |
 |
 |
|
| |
3.2.2.2.1.1 |
Pax-2, isoform 1 (long) |
|
 |
T00678 |
|
| |
3.2.2.2.1.2 |
Pax-2, isoform 2 (short) |
|
 |
T26232 |
|
| |
3.2.2.2.2 |
Pax-5 |
 |
 |
T01201 |
 |
 |
 |
| |
3.2.2.2.3 |
Pax-8 |
|
 |
T01828 |
 |
 |
 |
| |
3.3 |
Class: Fork head / winged helix factors |
 |
 |
| |
3.3.1 |
Family: Forkhead box (FOX) factors |
TGTTT |
|
| |
3.3.1.1 |
Subfamily: FOXA |
TGTTTGTTT |
|
| |
3.3.1.1.1 |
FOXA1 (HNF-3α) |
|
 |
T02343 |
 |
 |
|
| |
3.3.1.1.2 |
FOXA2 (HNF-3β) |
|
 |
T02344 |
 |
 |
|
| |
3.3.1.1.3 |
FOXA3 (HNF-3γ) |
|
 |
T02345 |
 |
 |
 |
| |
3.3.1.2 |
Subfamily: FOXB |
|
|
| |
3.3.1.2.1 |
FOXB1 (Fkh-5) |
 |
 |
T02443 |
 |
|
|
| |
3.3.1.2.2 |
FOXB2 |
|
 |
T02442 |
 |
|
|
| |
3.3.1.3 |
Subfamily: FOXC |
TGTTTATTTAC |
|
| |
3.3.1.3.1 |
FOXC1 (Fkh-L7, FREAC3) |
 |
 |
T02426 |
 |
 |
|
| |
3.3.1.3.2 |
FOXC2 (Fkh-L14) |
 |
 |
T02445 |
 |
 |
 |
| |
3.3.1.4 |
Subfamily: FOXD |
TGTTTACTTAAG |
|
| |
3.3.1.4.1 |
FOXD1 (Fkh-L8, FREAC4) |
|
 |
T02293 |
 |
 |
|
| |
3.3.1.4.2 |
FOXD2 (Fkh-L17, FREAC9) |
 |
 |
T02492 |
 |
 |
|
| |
3.3.1.4.3 |
FOXD3 (HFH-2) |
 |
 |
T04166 |
 |
 F F |
 |
| |
3.3.1.4.4 |
FOXD4 (Fkh-L9, FREAC5) |
|
 |
T02431 |
 |
 F F |
|
| |
3.3.1.5 |
Subfamily: FOXE |
|
|
| |
3.3.1.5.1 |
FOXE1 (FOXE2, Fkh-L15) |
|
 |
T06409 |
 |
 |
|
| |
3.3.1.5.2 |
FOXE3 (Fkh-L12, FREAC8) |
|
 |
T06410 |
 |
 |
|
| |
3.3.1.6 |
Subfamily: FOXF |
TGTTTATATG |
|
| |
3.3.1.6.1 |
FOXF1 (Fkh-L5, FREAC1) |
|
 |
T02461 |
 |
 |
|
| |
3.3.1.6.2 |
FOXF2 (Fkh-L6, FREAC2) |
|
 |
T02514 |
 |
|
|
| |
3.3.1.7 |
Subfamily: FOXG |
TTTGTTTTT |
|
| |
3.3.1.7.1 |
FOXG1 (Fkh-2) |
 |
 |
T02353 |
 |
 |
|
| |
3.3.1.8 |
Subfamily: FOXH |
TGTGTATT |
|
| |
3.3.1.8.1 |
FOXH1 |
|
 |
T09107 |
 |
 |
|
| |
3.3.1.8.1.1 |
FOXH1, isoform 1 (long) |
|
 |
T04100 |
|
| |
3.3.1.8.1.2 |
FOXH1, isoform 2 (short) |
|
 |
T |
|
| |
3.3.1.9 |
Subfamily: FOXI |
TGTTTGTTTA |
|
| |
3.3.1.9.1 |
FOXI1 (Fkh-L10, FREAC6) |
 |
 |
T02487 |
 |
 |
|
| |
3.3.1.9.2 |
FOXI2 |
|
 |
T09341 |
 |
 |
|
| |
3.3.1.9.3 |
FOXI3 |
|
 |
T |
 |
 |
|
| |
3.3.1.10 |
Subfamily: FOXJ |
TGTTTGTTTA |
|
| |
3.3.1.10.1 |
FOXJ1 (Fkh-L13, HFH-4) |
|
 |
T02460 |
 |
 |
|
| |
3.3.1.10.2 |
FOXJ2 (Fkx) |
 |
 |
T04170 |
 |
 |
|
| |
3.3.1.10.3 |
FOXJ3 (Fkx) |
 |
 |
T18933 |
 |
 |
|
| |
3.3.1.10.3.1 |
FOXJ3, isoform 1 |
|
 |
T18319 |
|
| |
3.3.1.10.3.2 |
FOXJ3, isoform 2 |
|
 |
T18320 |
|
| |
3.3.1.11 |
Subfamily: FOXK |
TTGTTTAC |
|
| |
3.3.1.11.1 |
FOXK1 |
 |
 |
T04216 |
 |
 |
|
| |
3.3.1.11.2 |
FOXK2 |
|
 |
T19940 |
 |
 F F |
|
| |
3.3.1.11.2.1 |
FOXK2, isoform 1 (FOXK2a) |
|
 |
T19939 |
|
| |
3.3.1.11.2.2 |
FOXK2, isoform 2 (FOXK2b) |
|
 |
T19941 |
|
| |
3.3.1.12 |
Subfamily: FOXL |
TGTTTATGT |
|
| |
3.3.1.12.1 |
FOXL1 (Fkh-L11, FREAC7) |
 |
 |
T02417 |
 |
|
|
| |
3.3.1.12.2 |
FOXL2 |
|
 |
T09367 |
 |
 |
|
| |
3.3.1.13 |
Subfamily: FOXM |
CTGTTTAT |
|
| |
3.3.1.13.1 |
FOXM1 (Fkh-L11, FREAC7) |
|
 |
T02515 |
 |
 |
|
| |
3.3.1.14 |
Subfamily: FOXN |
|
|
| |
3.3.1.14.1 |
FOXN1 (Whn) |
|
 |
T01835 |
 |
 F F |
|
| |
3.3.1.14.2 |
FOXN2 (HTLF) |
|
 |
T04205 |
 |
 |
|
| |
3.3.1.14.3 |
FOXN3 (CHES1) |
|
 |
T18939 |
 |
 |
|
| |
3.3.1.14.4 |
FOXN4 |
|
 |
T09463 |
 |
 |
|
| |
3.3.1.15 |
Subfamily: FOXO |
TTGTTTAC |
|
| |
3.3.1.15.1 |
FOXO1 (FKHR) |
 |
 |
T04203 |
 |
 |
|
| |
3.3.1.15.2 |
FOXO3 (FKHR-L1) |
 |
 |
T04178 |
 |
 |
|
| |
3.3.1.15.3 |
FOXO4 |
 |
 |
T04176 |
 |
 |
 |
| |
3.3.1.15.4 |
FOXO6 |
 |
 |
T19943 |
 |
 |
|
| |
3.3.1.16 |
Subfamily: FOXP |
TTGTTTCA |
|
| |
3.3.1.16.1 |
FOXP1 |
|
 |
T04815 |
 |
 |
|
| |
3.3.1.16.1.1 |
FOXP1, isoform 1 (A) |
|
 |
T02289 |
|
| |
3.3.1.16.1.2 |
FOXP1, isoform 2 (B) |
|
 |
T04812 |
|
| |
3.3.1.16.1.3 |
FOXP1, isoform 3 (C) |
|
 |
T04813 |
|
| |
3.3.1.16.1.4 |
FOXP1, isoform 4 (D) |
|
 |
T05943 |
|
| |
3.3.1.16.2 |
FOXP2 |
|
 |
T04814 |
 |
 |
 |
| |
3.3.1.16.2.1 |
FOXP2, isoform 1 |
|
 |
T14710 |
|
| |
3.3.1.16.2.2 |
FOXP2, isoform 2 |
|
 |
T26126 |
|
| |
3.3.1.16.3 |
FOXP3 |
 |
 |
T04279 |
 |
 |
|
| |
3.3.1.16.4 |
FOXP4 (FKHLA) |
|
 |
T11049 |
 |
 |
|
| |
3.3.1.16.4.1 |
FOXP4, isoform 1 |
|
 |
T05924 |
|
| |
3.3.1.16.4.2 |
FOXP4, isoform 2 |
|
 |
T18944 | |
| |
3.3.1.16.4.3 |
FOXP4, isoform 3 |
|
 |
T18945 |
|
| |
3.3.1.17 |
Subfamily: FOXQ |
|
|
| |
3.3.1.17.1 |
FOXQ1 (HFH-1) |
|
 |
T05952 |
 |
 |
|
| |
3.3.1.18 |
Subfamily: FOXR |
|
|
| |
3.3.1.18.1 |
FOXR1 |
|
 |
T19947 |
 |
 |
|
| |
3.3.1.18.1.1 |
FOXR1, isoform 1 |
|
 |
T19946 |
|
| |
3.3.1.18.1.2 |
FOXR1, isoform 2 |
|
 |
T19948 |
|
| |
3.3.1.18.2 |
FOXR2 |
|
 |
T19952 |
 |
 |
|
| |
3.3.1.19 |
Subfamily: FOXS |
|
|
| |
3.3.1.19.1 |
FOXS1 (Fkh-L18, FREAC10) |
|
 |
T02440 |
 |
 |
|
| |
3.3.2 |
Family: E2F-related factors |
|
|
| |
3.3.2.1 |
Subfamily: E2F |
|
|
| |
3.3.2.1.1 |
E2F-1 |
 |
 |
T01543 |
 |
 |
|
| |
3.3.2.1.2 |
E2F-2 |
 |
 |
T03526 |
 |
 |
|
| |
3.3.2.1.3 |
E2F-3 |
 |
 |
T09191 |
 |
 |
|
| |
3.3.2.1.4 |
E2F-4 |
 |
 |
T16329 |
 |
 |
 |
| |
3.3.2.1.5 |
E2F-5 |
|
 |
T02948 |
 |
 F F |
|
| |
3.3.2.1.6 |
E2F-6 |
|
 |
T03250 |
 |
 |
|
| |
3.3.2.1.7 |
E2F-7 |
 |
 |
T06112 |
 |
 |
|
| |
3.3.2.1.7.1 |
E2F-7, isoform 1 |
|
 |
T15104 |
|
| |
3.3.2.1.7.2 |
E2F-7, isoform 2 |
|
 |
T26851 |
|
| |
3.3.2.1.8 |
E2F-8 |
 |
 |
T |
 |
 |
|
| |
3.3.2.2 |
Subfamily: Dp-1 |
|
|
| |
3.3.2.2.1 |
Dp-1 (DRTF-1) |
|
 |
T01549 |
 |
|
|
| |
3.3.2.2.2 |
Dp-2 (Dp-3) |
|
 |
T04103 |
 |
 |
 |
| |
3.3.2.2.2.1 |
Dp-2α, isoform 1 |
|
 |
|
|
| |
3.3.2.2.2.2 |
Dp-2β, isoform 2 |
|
 |
|
|
| |
3.3.2.2.2.3 |
Dp-2γ, isoform 3 |
|
 |
|
|
| |
3.3.2.2.2.4 |
Dp-2δ, isoform 4 |
|
 |
|
|
| |
3.3.3 |
Family: RFX-related factors |
|
|
| |
3.3.3.0.1 |
RFX1 (EF-C) |
|
 |
T00231 |
 |
 |
 |
| |
3.3.3.0.2 |
RFX2 |
 |
 |
T01669 |
 |
 |
|
| |
3.3.3.0.2.1 |
RFX2, isoform 1 |
|
 |
T25991 |
|
| |
3.3.3.0.2.2 |
RFX2, isoform 2 |
|
 |
T15278 |
|
| |
3.3.3.0.3 |
RFX3 |
 |
 |
T01671 |
 |
 |
|
| |
3.3.3.0.3.1 |
RFX3, isoform 1 |
|
 |
T22177 |
|
| |
3.3.3.0.3.2 |
RFX3, isoform 2 |
|
 |
T26276 |
|
| |
3.3.3.0.4 |
RFX4 |
 |
 |
T19210 |
 |
 |
|
| |
3.3.3.0.4.1 |
RFX4, isoform 1 |
|
 |
T19208 |
|
| |
3.3.3.0.4.2 |
RFX4, isoform 2 |
|
 |
T19209 |
|
| |
3.3.3.0.4.3 |
RFX4, isoform 3 |
|
 |
T26277 |
|
| |
3.3.3.0.4.4 |
RFX4, isoform 4 |
|
 |
T19207 |
|
| |
3.3.3.0.5 |
RFX5 |
 |
 |
T05440 |
 |
 |
|
| |
3.3.3.0.6 |
RFX6 (RFXDC-1) |
|
 |
T |
 |
 |
|
| |
3.3.3.0.6.1 |
RFX6, isoform 1 |
|
 |
T |
|
| |
3.3.3.0.6.2 |
RFX6, isoform 2 |
|
 |
T |
|
| |
3.3.3.0.6.3 |
RFX6, isoform 3 |
|
 |
T |
|
| |
3.3.3.0.7 |
RFX7 (RFXDC-2) |
|
 |
T16909 |
 |
 |
|
| |
3.3.3.0.8 |
RFX8 |
|
 |
T |
 |
 |
|
| |
3.4 |
Class: Heat shock factors |
 |
 |
| |
3.4.1 |
Family: HSF factors |
TTCCAGAAGCTTC |
|
| |
3.4.1.0.1 |
HSF1 (HSTF1) |
 |
 |
T00384 |
 |
 |
 |
| |
3.4.1.0.1.1 |
HSF1, isoform 1 |
|
 |
T05813 |
|
| |
3.4.1.0.1.2 |
HSF1, isoform 2 |
|
 |
T01525 |
|
| |
3.4.1.0.2 |
HSF2 (HSTF2) |
 |
 |
T00972 |
 |
 |
|
| |
3.4.1.0.2.1 |
HSF2, isoform α |
|
 |
T25984 |
|
| |
3.4.1.0.2.2 |
HSF2, isoform β |
|
 |
T08289 |
|
| |
3.4.1.0.3 |
HSF4 (HSTF4) |
 |
 |
T05809 |
 |
 |
|
| |
3.4.1.0.3.1 |
HSF4, isoform A |
|
 |
T05810 |
|
| |
3.4.1.0.3.1 |
HSF4, isoform B |
|
 |
T05811 |
|
| |
3.4.1.0.4 |
HSF5 (HSTF5) |
|
 |
T19994 |
 |
 F F |
|
| |
3.4.1.0.6 |
HSFY1 (HSFY2, HSF2L) |
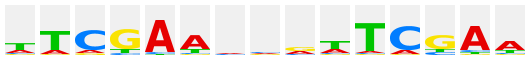 |
 |
T |
 |
 |
|
| |
3.4.1.0.7 |
HSF3 |
|
 |
T |
|
 |
|
| |
3.4.1.0.7.1 |
HSF3, isoform 1 (a) |
|
 |
|
|
| |
3.4.1.0.7.2 |
HSF3, isoform 2 (b) |
|
 |
|
|
| |
3.5 |
Class: Tryptophan cluster factors |
 |
 |
| |
3.5.1 |
Family: Myb/SANT domain factors |
|
|
| |
3.5.1.1 |
Subfamily: Myb-like factors |
CAGTTG |
|
| |
3.5.1.1.1 |
c-Myb |
|
 |
T00138 |
 |
 |
 |
| |
3.5.1.1.2 |
A-Myb (Myb-L1) |
 |
 |
T22728 |
 |
 |
|
| |
3.5.1.1.2.1 |
A-Myb, isoform 1 (long) |
|
 |
T01584 |
|
| |
3.5.1.1.2.2 |
A-Myb, isoform 2 (short) |
|
 |
T25953 |
|
| |
3.5.1.1.3 |
B-Myb (Myb-L2) |
 |
 |
T01587 |
 |
 |
 |
| |
3.5.1.1.4 |
CDC5L |
|
 |
T18831 |
 |
 |
|
| |
3.5.1.1.5 |
SNAPc4 (PTFα) |
|
 |
T19242 |
 |
 |
|
| |
3.5.1.1.5.1 |
SNAPc4, isoform 1 |
|
 |
T25992 |
|
| |
3.5.1.1.5.2 |
SNAPc4, isoform 2 |
|
 |
T25993 |
|
| |
3.5.1.1.5.3 |
SNAPc4, isoform 3 |
|
 |
T25994 |
|
| |
3.5.1.2 |
Subfamily: NCoR-like factors |
|
|
| |
3.5.1.2.1 |
NCoR1 |
|
 |
T04688 |
 |
 |
|
| |
3.5.1.2.1.1 |
NCoR1, isoform 1 |
|
 |
T08641 |
|
| |
3.5.1.2.1.2 |
NCoR1, isoform 2 |
|
 |
T19110 |
|
| |
3.5.1.2.2 |
NCoR2 |
|
 |
T04690 |
 |
 |
|
| |
3.5.1.2.2.1 |
NCoR2, isoform α |
|
 |
T09272 |
|
| |
3.5.1.2.2.2 |
NCoR2, isoform β |
|
 |
T19114 |
|
| |
3.5.1.2.3 |
RCoR1 |
|
 |
T |
 |
 F F |
|
| |
3.5.1.2.4 |
RCoR2 |
|
 |
T |
 |
 |
|
| |
3.5.1.2.5 |
RCoR3 |
|
 |
T27833 |
 |
 |
|
| |
3.5.1.2.5.1 |
RCoR3, isoform 1 |
|
 |
T27886 |
|
| |
3.5.1.2.5.2 |
RCoR3, isoform 2 |
|
 |
T27386 |
|
| |
3.5.1.3 |
Subfamily: MIER-like factors |
|
|
| |
3.5.1.3.1 |
MIER1 |
|
 |
T27845 |
 |
 |
|
| |
3.5.1.3.1.1 |
MIER1, isoform 1 (N4-β) |
|
 |
T27923 |
|
| |
3.5.1.3.1.2 |
MIER1, isoform 2 (N1-β) |
|
 |
T27349 |
|
| |
3.5.1.3.1.3 |
MIER1, isoform 3 (N3-β) |
|
 |
T27348 |
|
| |
3.5.1.3.1.4 |
MIER1, isoform 4 (N2-β) |
|
 |
T27924 |
|
| |
3.5.1.3.1.5 |
MIER1, isoform 5 |
|
 |
T27925 |
|
| |
3.5.1.3.2 |
MIER2 |
|
 |
T20020 |
 |
 |
|
| |
3.5.1.3.2.1 |
MIER2, isoform 1 |
|
 |
T20019 |
|
| |
3.5.1.3.2.2 |
MIER2, isoform 2 |
|
 |
T20021 |
|
| |
3.5.1.3.2.3 |
MIER2, isoform 3 |
|
 |
T20022 |
|
| |
3.5.1.3.3 |
MIER3 |
|
 |
T27846 |
 |
 |
|
| |
3.5.1.3.3.1 |
MIER3, isoform 1 |
|
 |
T27350 |
|
| |
3.5.1.3.3.2 |
MIER3, isoform 2 |
|
 |
T27926 |
|
| |
3.5.1.3.4 |
MTA1 = 2.2.1.2.5 |
|
 |
T27821 |
 |
 |
|
| |
3.5.1.3.5 |
MTA2 (MTA1-L1) = 2.2.1.2.6 |
|
 |
T |
 |
 |
|
| |
3.5.1.3.6 |
MTA3 = 2.2.1.2.7 |
|
 |
T27832 |
 |
 F F |
|
| |
3.5.1.3.6.1 |
MTA3, isoform 1 |
|
 |
T27227 |
|
| |
3.5.1.3.6.2 |
MTA3, isoform 2 |
|
 |
T27228 |
|
| |
3.5.1.3.7 |
RERE (ARG, ARP, ATN1L) = 2.2.1.2.4 |
|
 |
T |
 |
 |
|
| |
3.5.1.3.8 |
DMAP1 |
|
 |
T |
 |
 |
|
| |
3.5.1.3.8.1 |
DMAP1, isoform 1 |
|
 |
T |
|
| |
3.5.1.3.8.2 |
DMAP1, isoform 2 |
|
 |
T |
|
| |
3.5.1.3.9 |
TRERF1 (Rapa, TReP-132) = 2.3.4.20.1 |
|
 |
T16489 |
 |
 |
|
| |
3.5.1.3.9.1 |
TRERF1, isoform 1 |
|
 |
T16488 |
|
| |
3.5.1.3.9.2 |
TRERF1, isoform 2 |
|
 |
T16490 |
|
| |
3.5.1.3.9.3 |
TRERF1, isoform 3 [1] |
|
 |
T16491 |
|
| |
3.5.1.3.9.4 |
TRERF1, isoform 4 |
|
 |
T16492 |
|
| |
3.5.1.3.10 |
ZNF541 = 2.3.4.20.2 |
|
 |
T |
 |
 |
|
| |
3.5.1.3.10.1 |
ZNF541, isoform 1 |
|
 |
T |
|
| |
3.5.1.3.10.2 |
ZNF541, isoform 2 |
|
 |
T |
|
| |
3.5.1.3.11 |
CRAMP1L (HN1L) |
|
 |
T |
 |
 |
|
| |
3.5.1.3.12 |
ELMSAN1 (C14orf43) |
|
 |
T |
 |
 |
|
| |
3.5.1.4 |
Subfamily: DNAJC-like factors |
|
|
| |
3.5.1.4.1 |
DNAJC1 |
|
 |
T |
 |
 |
|
| |
3.5.1.4.2 |
DNAJC2 (ZRF1) |
|
 |
T27836 |
 |
 |
|
| |
3.5.1.5 |
Subfamily: SMARCA-like factors |
|
|
| |
3.5.1.5.1 |
SMARCA1 (SNF2L) |
|
 |
T20112 |
 |
 |
|
| |
3.5.1.5.1.1 |
SMARCA1, isoform 1 |
|
 |
T20111 |
|
| |
3.5.1.5.1.2 |
SMARCA1, isoform 2 |
|
 |
T20113 |
|
| |
3.5.1.5.2 |
SMARCA5 (SNF2H) |
|
 |
T |
 |
 |
|
| |
3.5.1.6 |
Subfamily: TADA2-like factors |
|
|
| |
3.5.1.6.1 |
TAD-2α (TADA2A, ADA2-like) |
|
 |
T19286 |
 |
 |
 |
| |
3.5.1.6.2 |
TAD-2β (TADA2B, ADA2-β) |
|
 |
T |
 |
 |
|
| |
3.5.1.6.3 |
SMARCC1 (BAF155) |
|
 |
T |
 |
 |
|
| |
3.5.1.6.3.1 |
SMARCC1, isoform 1 |
|
 |
T |
|
| |
3.5.1.6.3.2 |
SMARCC1, isoform 2 |
|
 |
T |
|
| |
3.5.1.6.4 |
SMARCC2 (BAF170) |
|
 |
T |
 |
 |
|
| |
3.5.1.6.4.1 |
SMARCC2, isoform 1 |
|
 |
T |
|
| |
3.5.1.6.4.2 |
SMARCC2, isoform 2 |
|
 |
T |
|
| |
3.5.1.7 |
Subfamily: DMTF-like factors |
CCCGGATGT |
|
| |
3.5.1.7.1 |
DMTF1 |
|
 |
T19913 |
 |
 |
|
| |
3.5.1.7.1.1 |
DMTF1, isoform 1 |
|
 |
T19912 |
|
| |
3.5.1.7.1.2 |
DMTF1, isoform 2 |
|
 |
T19915 |
|
| |
3.5.1.7.1.3 |
DMTF1, isoform 3 |
|
 |
T19916 |
|
| |
3.5.1.7.1.4 |
DMTF1, isoform 4 |
|
 |
T19917 |
|
| |
3.5.1.7.1.5 |
DMTF1, isoform 5 |
|
 |
T19914 |
|
| |
3.5.2 |
Family: Ets-related factors |
GGAAG |
|
| |
3.5.2.1 |
Subfamily: Ets-like factors |
CCGGAAG |
|
| |
3.5.2.1.1 |
c-Ets-1 |
 |
 |
T00111 |
 |
 |
 |
| |
3.5.2.1.2 |
c-Ets-2 |
|
 |
T09489 |
 |
 |
|
| |
3.5.2.1.3 |
ETV2 |
 |
 |
T15697 |
 |
 |
|
| |
3.5.2.1.4 |
GABPα (E4TF1-60, NRSF-2α) |
 |
 |
T01402 |
 |
 |
|
| |
3.5.2.1.5 |
Fli-1 |
 |
 |
T01408 |
 |
 |
|
| |
3.5.2.1.6 |
ERG |
 |
 |
T17954 |
 |
 |
|
| |
3.5.2.1.6.1 |
ERG, isoform 1 |
|
 |
T17955 |
|
| |
3.5.2.1.6.2 |
ERG, isoform 2 |
|
 |
T17956 |
|
| |
3.5.2.1.6.3 |
ERG, isoform 3 |
|
 |
T17953 |
|
| |
3.5.2.1.6.4 |
ERG, isoform 5 |
|
 |
T17957 |
|
| |
3.5.2.1.7 |
FEV (Pet-1) |
 |
 |
T05431 |
 |
 |
|
| |
3.5.2.1.8 |
ETV3 (PE-1) |
 |
 |
T18918 |
 |
 |
|
| |
3.5.2.1.10 |
ERF |
 |
 |
T16656 |
 |
 F F |
|
| |
3.5.2.2 |
Subfamily: Elk-like factors |
CCGGAAG |
|
| |
3.5.2.2.1 |
Elk-1 |
 |
 |
T05013 |
 |
 |
 |
| |
3.5.2.2.2 |
Elk-3 (SAP-2) |
 |
 |
T09982 |
 |
 |
|
| |
3.5.2.2.3 |
Elk-4 (SAP-1) |
 |
 |
T06074 |
 |
 |
 |
| |
3.5.2.2.4 |
ETV1 |
 |
 |
T08214 |
 |
 |
 |
| |
3.5.2.2.5 |
ETV4 (E1A-F) |
 |
 |
T00684 |
 |
 |
|
| |
3.5.2.2.5.1 |
ETV4, isoform 1 |
|
 |
T09992 |
|
| |
3.5.2.2.5.2 |
ETV4, isoform 2 |
|
 |
T30270 |
|
| |
3.5.2.2.6 |
ETV5 |
 |
 |
T08200 |
 |
 |
|
| |
3.5.2.3 |
Subfamily: Elf-1-like factors |
CCGGAAAT |
|
| |
3.5.2.3.1 |
Elf-1 |
 |
 |
T05012 |
 |
 |
|
| |
3.5.2.3.2 |
Elf-2 (NERF) |
|
 |
T16604 |
 |
 |
|
| |
3.5.2.3.2.1 |
Elf-2, isoform 1 (Elf-2a) |
|
 |
T18898 |
|
| |
3.5.2.3.2.2 |
Elf-2, isoform 2 (Elf-2b) |
|
 |
T18899 |
|
| |
3.5.2.3.2.3 |
Elf-2, isoform 3 |
|
 |
T18900 |
|
| |
3.5.2.3.2.4 |
Elf-2, isoform 4 |
|
 |
T18897 |
|
| |
3.5.2.3.3 |
Elf-4 (ELFR, MEF) |
 |
 |
T21528 |
 |
 |
|
| |
3.5.2.4 |
Subfamily: EHF-like factors |
CCGGAAGT |
|
| |
3.5.2.4.1 |
EHF (ESE-3) |
 |
 |
T17678 |
 |
 |
|
| |
3.5.2.4.1.1 |
EHF, isoform 1 |
|
 |
T17903 |
|
| |
3.5.2.4.1.2 |
EHF, isoform 2 |
|
 |
T26095 |
|
| |
3.5.2.4.1.3 |
EHF, isoform 3 |
|
 |
T26096 |
|
| |
3.5.2.4.2 |
Elf-3 (ESE-1) |
 |
 |
T08074 |
 |
 |
 |
| |
3.5.2.4.2.1 |
Elf-3, isoform 1 |
|
 |
T25982 |
|
| |
3.5.2.4.2.2 |
Elf-3, isoform 2 |
|
 |
T15555 |
|
| |
3.5.2.4.3 |
Elf-5 (ESE-2) |
 |
 |
T15298 |
 |
 |
|
| |
3.5.2.5 |
Subfamily: Spi-like factors |
AGAGGAAG |
|
| |
3.5.2.5.1 |
PU.1 (Spi-1) |
 |
 |
T00702 |
 |
 |
 |
| |
3.5.2.5.2 |
Spi-B |
 |
 |
T14146 |
 |
 |
|
| |
3.5.2.5.2.1 |
Spi-B, isoform IA |
|
 |
T19268 |
|
| |
3.5.2.5.2.2 |
Spi-B, isoform IC |
|
 |
T26304 |
|
| |
3.5.2.5.2.3 |
Spi-B, isoform IIA |
|
 |
T19269 |
|
| |
3.5.2.5.3 |
Spi-C |
 |
 |
T20136 |
 |
 |
|
| |
3.5.2.5.3.1 |
Spi-C, isoform 1 |
|
 |
T20135 |
|
| |
3.5.2.5.3.2 |
Spi-C, isoform 2 |
|
 |
T20137 |
|
| |
3.5.2.6 |
Subfamily: ETV6-like factors |
CCCGGAAGT |
|
| |
3.5.2.6.1 |
ETV6 (Tel-1) |
 |
 |
T08872 |
 |
 |
|
| |
3.5.2.7 |
Subfamily: SPDEF-like factors |
CCGGATGT |
|
| |
3.5.2.7.1 |
SPDEF (PSE) |
 |
 |
T05406 |
 |
 |
 |
| |
3.5.3 |
Family: Interferon-regulatory factors |
AAGTGAA |
|
| |
3.5.3.0.1 |
IRF-1 |
|
 |
T00422 |
 |
 |
 |
| |
3.5.3.0.2 |
IRF-2 |
|
 |
T00425 |
 |
 |
 |
| |
3.5.3.0.3 |
IRF-3 |
 |
 |
T05128 |
 |
 |
 |
| |
3.5.3.0.4 |
IRF-4 (LSIRF, NF-EM5, MUM1, Pip) |
 |
 |
T01412 |
 |
 |
|
| |
3.5.3.0.4.1 |
IRF-4, isoform 1 |
|
 |
T09278 |
|
| |
3.5.3.0.4.2 |
IRF-4, isoform 2 |
|
 |
T21769 |
|
| |
3.5.3.0.5 |
IRF-5 |
 |
 |
T05103 |
 |
 |
|
| |
3.5.3.0.6 |
IRF-6 |
|
 |
T05119 |
 |
 |
|
| |
3.5.3.0.7 |
IRF-7 |
 |
 |
T05144 |
 |
 |
 |
| |
3.5.3.0.8 |
IRF-8 (ICSBP1) |
 |
 |
T00402 |
 |
 |
|
| |
3.5.3.0.9 |
IRF-9 (ISGF-3γ) |
 |
 |
T05096 |
 |
 |
|
| |
3.6 |
Class: TEA domain factors |
 |
 |
| |
3.6.1 |
Family: TEF-1-related factors |
CATTCC |
|
| |
3.6.1.0.1 |
TEF-1 (TEAD-1, TCF-13) |
 |
 |
T01124 |
 |
 |
 |
| |
3.6.1.0.2 |
TEF-3 (TEAD-4, TCF-13L1, Tefr1) |
 |
 |
T19305 |
 |
 |
|
| |
3.6.1.0.2.1 |
TEF-3, isoform 1 (TEFR1A, ETFR-2A) |
|
 |
T19303 |
|
| |
3.6.1.0.2.2 |
TEF-3, isoform 2 (TEFR1B, ETFR-2B) |
|
 |
T19304 |
|
| |
3.6.1.0.3 |
TEF-4 (TEAD-2) |
|
 |
T09525 |
 |
 |
 |
| |
3.6.1.0.4 |
TEF-5 (TEAD-3, TEAD5) |
 |
 |
T19302 |
 |
 |
|
| |
3.7 |
Class: ARID domain factors |

 |
 |
| |
3.7.1 |
Family: ARID-related factors |
|
|
| |
3.7.1.1 |
Subfamily: ARID1 |
|
|
| |
3.7.1.1.1 |
ARID1A (BAF250, BAF250A, OSA1, SMARCF1) |
|
 |
T27839 |
 |
 |
|
| |
3.7.1.1.1.1 |
ARID1A, isoform 1 |
|
 |
T27259 |
|
| |
3.7.1.1.1.2 |
ARID1A, isoform 2 |
|
 |
T27260 |
|
| |
3.7.1.1.1.3 |
ARID1A, isoform 3 |
|
 |
T27261 |
|
| |
3.7.1.1.1.4 |
ARID1A, isoform 4 |
|
 |
T27891 |
|
| |
3.7.1.1.2 |
ARID1B (BAF250, BAF250A, OSA1, SMARCF1) |
|
 |
T |
 |
 F F |
 |
| |
3.7.1.2 |
Subfamily: ARID2 |
|
|
| |
3.7.1.2.1 |
ARID2 (BAF200) |
|
 |
T |
 |
 |
|
| |
3.7.1.3 |
Subfamily: ARID3 |
|
|
| |
3.7.1.3.1 |
ARID3A (Bright, DRIL1, DRIL3, DRX, E2FBP1) |
|
 |
T06432 |
 |
 |
|
| |
3.7.1.3.2 |
ARID3B (BDP, DRIL2) |
|
 |
T06433 |
 |
 |
|
| |
3.7.1.3.2.1 |
ARID3B, isoform 1 |
|
 |
T14720 |
|
| |
3.7.1.3.2.2 |
ARID3B, isoform 2 |
|
 |
T26024 |
|
| |
3.7.1.3.2.3 |
ARID3B, isoform 3 |
|
 |
T26025 |
|
| |
3.7.1.3.3 |
ARID3C |
|
 |
T19835 |
 |
 |
|
| |
3.7.1.4 |
Subfamily: ARID4 |
|
|
| |
3.7.1.4.1 |
ARID4A (RBBP1, RBP1) |
|
 |
T |
 |
 |
|
| |
3.7.1.4.2 |
ARID4B (BRCAA1, RBBP1L1, RBP1L1, SAP180) |
|
 |
T18778 |
 |
 |
|
| |
3.7.1.4.2.1 |
ARID4B, isoform 1 |
|
 |
T06439 |
|
| |
3.7.1.4.2.2 |
ARID4B, isoform 2 |
|
 |
T18780 |
|
| |
3.7.1.5 |
Subfamily: ARID5 |
|
|
| |
3.7.1.5.1 |
ARID5A (MRF1) |
|
 |
T16757 |
 |
 |
|
| |
3.7.1.5.1.1 |
ARID5A, isoform 1 |
|
 |
T16756 |
|
| |
3.7.1.5.1.2 |
ARID5A, isoform 2 |
|
 |
T16758 |
|
| |
3.7.1.5.1.3 |
ARID5A, isoform 3 |
|
 |
T16761 |
|
| |
3.7.1.5.1.4 |
ARID5A, isoform 4 |
|
 |
T16764 |
|
| |
3.7.1.5.1.5 |
ARID5A, isoform 5 |
|
 |
T16765 |
|
| |
3.7.1.5.2 |
ARID5B (DESRT, MRF2) |
|
 |
T11231 |
 |
 |
 |
| |
3.7.1.5.2.1 |
ARID5B, isoform 1 |
|
 |
T06464 |
|
| |
3.7.1.5.2.2 |
ARID5B, isoform 2 |
|
 |
T06463 |
|
| |
3.7.1.5.2.3 |
ARID5B, isoform 3 |
|
 |
T26027 |
|
| |
3.7.1.6 |
Subfamily: JARID1 |
|
|
| |
3.7.1.6.1 |
JARID1A (KDM5A, RBBP2, RBP2) |
|
 |
T |
 |
 F F |
|
| |
3.7.1.6.2 |
JARID1B (KDM5B, PLU1, RBBP2H1) |
|
 |
T27844 |
 |
 |
 |
| |
3.7.1.6.2.1 |
JARID1B, isoform 1 |
|
 |
T27347 |
|
| |
3.7.1.6.2.2 |
JARID1B, isoform 2 |
|
 |
T27922 |
|
| |
3.7.1.6.3 |
JARID1C (KDM5C, DXS1272E, SMCX, XE169) |
|
 |
T |
 |
 |
|
| |
3.7.1.6.3.1 |
JARID1C, isoform 1 |
|
 |
T |
|
| |
3.7.1.6.3.2 |
JARID1C, isoform 2 |
|
 |
T |
|
| |
3.7.1.6.3.3 |
JARID1C, isoform 3 |
|
 |
T |
|
| |
3.7.1.6.4 |
JARID1D (KDM5D, HY, HYA, SMCY) |
|
 |
T |
 |
 |
 |
| |
3.7.1.6.4.1 |
JARID1D, isoform 1 |
|
 |
T |
|
| |
3.7.1.6.4.2 |
JARID1D, isoform 2 |
|
 |
T |
|
| |
3.7.1.6.4.3 |
JARID1D, isoform 3 |
|
 |
T |
|
| |
3.7.1.6.4.4 |
JARID1D, isoform 4 |
|
 |
T |
|
| |
3.7.1.7 |
Subfamily: JARID2 |
|
|
| |
3.7.1.7.1 |
JARID2 (Jumonji, JMJ) |
|
 |
T |
 |
 |
 |
| |
3.7.1.7.1.1 |
JARID2, isoform 1 |
|
 |
T |
|
| |
3.7.1.7.1.2 |
JARID2, isoform 2 |
|
 |
T |
|
|
| 4 |
Superclass: Other all-α-helical DNA-binding domains |
 |
| |
4.1 |
Class: High-mobility group (HMG) domain factors |
 |
 |
| |
4.1.1 |
Family: SOX-related factors |
ACAA |
|
| |
4.1.1.1 |
Subfamily: Group A |
AAACAAA |
|
| |
4.1.1.1.1 |
SRY (TDF) |
 |
 |
T00996 |
 |
|
 |
| |
4.1.1.2 |
Subfamily: Group B |
AACAAAG |
|
| |
4.1.1.2.1 |
SOX-1 |
 |
 |
T06025 |
 |
|
|
| |
4.1.1.2.2 |
SOX-2 |
 |
 |
T01836 |
 |
|
 |
| |
4.1.1.2.3 |
SOX-3 |
 |
 |
T06026 |
 |
 F F |
|
| |
4.1.1.2.4 |
SOX-14 (SOX-28) |
 |
 |
T16939 |
 |
 |
|
| |
4.1.1.2.5 |
SOX-21 |
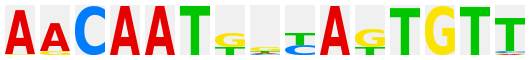 |
 |
T16926 |
 |
|
|
| |
4.1.1.3 |
Subfamily: Group C |
AACAAAGG |
|
| |
4.1.1.3.1 |
SOX-4 (IRE-ABP) |
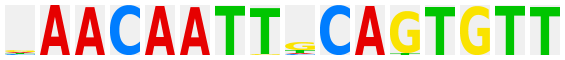 |
 |
T17807 |
 |
|
 |
| |
4.1.1.3.2 |
SOX-11 |
 |
 |
T17001 |
 |
 |
|
| |
4.1.1.3.3 |
SOX-12 (SOX-22) |
|
 |
T16937 |
 |
 |
|
| |
4.1.1.4 |
Subfamily: Group D |
AACAAT |
|
| |
4.1.1.4.1 |
SOX-5 |
|
 |
T01429 |
 |
 |
 |
| |
4.1.1.4.1.1 |
SOX-5A, isoform 1 |
|
 |
|
|
| |
4.1.1.4.1.2 |
SOX-5B, isoform 2 (L-Sox5) |
|
 |
T18021 |
|
| |
4.1.1.4.1.3 |
SOX-5, isoform 3 |
|
 |
T26301 |
|
| |
4.1.1.4.2 |
SOX-6 |
|
 |
T09432 |
 |
 |
|
| |
4.1.1.4.2.1 |
SOX-6, isoform 1 |
|
 |
T02897 |
|
| |
4.1.1.4.2.2 |
SOX-6, isoform 2 |
|
 |
T01844 |
|
| |
4.1.1.4.3 |
SOX-13 |
|
 |
T02420 |
 |
 |
|
| |
4.1.1.5 |
Subfamily: Group E |
AAACAAAGG |
|
| |
4.1.1.5.1 |
SOX-8 |
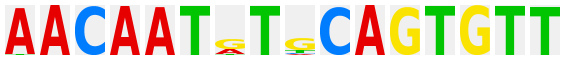 |
 |
T19255 |
 |
 |
|
| |
4.1.1.5.2 |
SOX-9 |
 |
 |
T01854 |
 |
 |
 |
| |
4.1.1.5.3 |
SOX-10 |
 |
 |
T08336 |
 |
 |
|
| |
4.1.1.6 |
Subfamily: Group F |
AACAAT |
|
| |
4.1.1.6.1 |
SOX-7 |
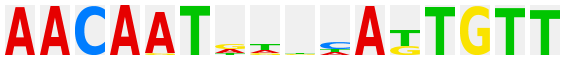 |
 |
T06032 |
 |
 |
|
| |
4.1.1.6.2 |
SOX-17 |
 |
 |
T06029 |
 |
 |
 |
| |
4.1.1.6.2.1 |
SOX-17, isoform 1 |
|
 |
T17872 |
|
| |
4.1.1.6.2.2 |
SOX-17, isoform 2 (T-SOX17) |
|
 |
T19249 |
|
| |
4.1.1.6.3 |
SOX-18 |
 |
 |
T01843 |
 |
 |
|
| |
4.1.1.7 |
Subfamily: Group G |
|
|
| |
4.1.1.7.1 |
SOX-15 |
 |
 |
T02896 |
 |
 |
|
| |
4.1.1.8 |
Subfamily: Group H |
GGACAATGG |
|
| |
4.1.1.8.1 |
SOX-30 |
|
 |
T19254 |
 |
 |
|
| |
4.1.1.9 |
Subfamily: Further Sox-related factors |
TTCATTCAA |
|
| |
4.1.1.9.1 |
CIC [1] |
|
 |
T19878 |
 |
 |
|
| |
4.1.1.9.1.1 |
CIC, isoform 1 (L) |
|
 |
T26532 |
|
| |
4.1.1.9.1.2 |
CIC, isoform 2 (S) |
|
 |
T19880 |
|
| |
4.1.1.9.2 |
HBP-1 [1] |
|
 |
T16354 |
 |
 |
|
| |
4.1.1.9.2.1 |
HBP-1, isoform 1 |
|
 |
T17384 |
|
| |
4.1.1.9.2.2 |
HBP-1, isoform 2 |
|
 |
T17385 |
|
| |
4.1.1.9.3 |
BBX (HBP-2) [1] |
|
 |
T16783 |
 |
 |
 |
| |
4.1.1.9.3.1 |
BBX, isoform 1 |
|
 |
T16781 |
|
| |
4.1.1.9.3.2 |
BBX, isoform 2 |
|
 |
T16786 |
|
| |
4.1.1.9.3.3 |
BBX, isoform 3 |
|
 |
T16787 |
|
| |
4.1.1.9.3.4 |
BBX, isoform 4 |
|
 |
T16789 |
|
| |
4.1.2 |
Family: TOX-related factors |
|
|
| |
4.1.2.0.1 |
TOX [1] |
|
 |
T |
 |
 |
 |
| |
4.1.2.0.1.1 |
TOX, isoform 1 |
|
 |
T |
|
| |
4.1.2.0.1.2 |
TOX, isoform 2 |
|
 |
T |
|
| |
4.1.2.0.2 |
TOX-2 (GCX-1) [1] |
|
 |
T |
 |
 |
|
| |
4.1.2.0.3 |
TOX-3 (CAGF9) [1] |
|
 |
T |
 |
 |
|
| |
4.1.2.0.4 |
TOX-4 (LCP-1) [1] |
|
 |
T |
 |
 |
|
| |
4.1.2.0.5 |
SMARCE1 [1] |
|
 |
T27820 |
 |
 |
|
| |
4.1.2.0.6 |
HMG20A (HMGX1) [1] |
|
 |
T18998 |
 |
 |
|
| |
4.1.2.0.6.1 |
HMG20A, isoform 1 |
|
 |
T18994 |
|
| |
4.1.2.0.6.2 |
HMG20A, isoform 2 |
|
 |
T18995 |
|
| |
4.1.2.0.6.3 |
HMG20A, isoform 3 |
|
 |
T18996 |
|
| |
4.1.2.0.6.4 |
HMG20A, isoform 4 |
|
 |
T18997 |
|
| |
4.1.2.0.7 |
HMG20B (HMGX2) [1] |
|
 |
T19003 |
 |
 |
 |
| |
4.1.2.0.8 |
HMGXB3 (SMF) [1] |
|
 |
T |
 |
 |
|
| |
4.1.3 |
Family: TCF-7-related factors |
TTCAAAG |
|
| |
4.1.3.0.1 |
TCF-7 (TCF-1) [1] |
 |
 |
T11358 |
 |
 |
|
| |
4.1.3.0.1.1 |
TCF-7, isoform 1 |
|
 |
T01109 |
|
| |
4.1.3.0.1.2 |
TCF-7, isoform 2 |
|
 |
T02945 |
|
| |
4.1.3.0.2 |
TCF-7L1 (TCF-3) [1] |
 |
 |
T01111 |
 |
 |
|
| |
4.1.3.0.3 |
TCF-7L2 (TCF-4) [1] |
|
 |
T08958 |
 |
 F F |
|
| |
4.1.3.0.3.1 |
TCF-7L2, isoform 1 |
|
 |
T02915 |
|
| |
4.1.3.0.3.2 |
TCF-7L2, isoform 2 |
|
 |
T02913 |
|
| |
4.1.3.0.3.3 |
TCF-7L2, isoform 3 |
|
 |
T19328 |
|
| |
4.1.3.0.3.4 |
TCF-7L2, isoform 4 |
|
 |
T19325 |
|
| |
4.1.3.0.3.5 |
TCF-7L2, isoform 5 |
|
 |
T19329 |
|
| |
4.1.3.0.3.6 |
TCF-7L2, isoform 6 |
|
 |
T19326 |
|
| |
4.1.3.0.3.7 |
TCF-7L2, isoform 7 |
|
 |
T19330 |
|
| |
4.1.3.0.3.8 |
TCF-7L2, isoform 8 |
|
 |
T19327 |
|
| |
4.1.3.0.3.9 |
TCF-7L2, isoform 9 |
|
 |
T26493 |
|
| |
4.1.3.0.4 |
LEF-1 (TCF-1α) [1] |
|
 |
T09576 |
 |
 |
 |
| |
4.1.3.0.4.1 |
LEF-1, isoform 1 |
|
 |
T00930 |
|
| |
4.1.3.0.4.2 |
LEF-1, isoform 2 (LEF-1S) |
|
 |
T01389 |
|
| |
4.1.3.0.5 |
TAF-1 (TAF-2A, TAF(II)250) [1] |
|
 |
T |
 |
 |
|
| |
4.1.3.0.5.1 |
TAF-1, isoform 1 |
|
 |
T |
|
| |
4.1.3.0.5.2 |
TAF-1, isoform 2 |
|
 |
T |
|
| |
4.1.3.0.5.3 |
TAF-1, isoform 3 |
|
 |
T |
|
| |
4.1.4 |
Family: PBRM1-related factors |
|
|
| |
4.1.4.0.1 |
PB-1 (PBRM1, BAF180) [1] |
|
 |
T21459 |
 |
 |
|
| |
4.1.4.0.1.1 |
PB-1, isoform 1 |
|
 |
T21458 |
|
| |
4.1.4.0.1.2 |
PB-1, isoform 2 |
|
 |
T21460 |
|
| |
4.1.4.0.2 |
HMGXB4 (HMG2L1) [1] |
|
 |
T |
 |
 |
|
| |
4.1.5 |
Family: WHSC1-related factors |
GGCATTGGAAA |
|
| |
4.1.5.0.1 |
WHSC1 (TRX-5) [1] |
|
 |
T19161 |
 |
 |
|
| |
4.1.5.0.1.1 |
WHSC1, isoform 1 |
|
 |
T19160 |
|
| |
4.1.5.0.1.2 |
WHSC1, isoform 2 |
|
 |
T19159 |
|
| |
4.1.5.0.1.3 |
WHSC1, isoform 3 |
|
 |
T19162 |
|
| |
4.1.5.0.1.4 |
WHSC1, isoform 4 (RE-IIBP) |
|
 |
T26863 |
|
| |
4.1.5.0.2 |
SSRP1 [1] |
|
 |
T25655 |
 |
 |
|
| |
4.1.5.0.2.1 |
SSRP1, isoform 1 |
|
 |
T01856 |
|
| |
4.1.5.0.2.2 |
SSRP1, isoform 2 |
|
 |
T26310 |
|
| |
4.1.6 |
Family: UBF-related factors |
|
|
| |
4.1.6.1 |
Subfamily: UBF-related factors |
|
|
| |
4.1.6.1.1 |
UBF [6] |
|
 |
T00860 |
 |
 |
 ; ; |
| |
4.1.6.1.1.1 |
UBF1 |
|
 |
T11305 |
|
| |
4.1.6.1.1.2 |
UBF2 |
|
 |
T11304 |
|
| |
4.1.6.2 |
Subfamily: UBF-like proteins |
|
|
| |
4.1.6.2.1 |
UBTFL1 [2] |
|
 |
T |
 |
 |
|
| |
4.1.7 |
Family: TFAM |
|
|
| |
4.1.7.0.1 |
TFAM (mtTFA, MtTF1, TCF6, TCF6L2) [2] |
|
 |
T14300 |
 |
 |
 |
| |
4.1.7.0.1.1 |
TFAM, isoform 1 (mitochondrial) |
|
 |
T14299 |
|
| |
4.1.7.0.1.2 |
TFAM, isoform 2 (nuclear) |
|
 |
T14301 |
|
| |
4.2 |
Class: Heteromeric CCAAT-binding factors |
 |
| |
4.2.1 |
Family: Heteromeric CCAAT-binding factors |
CCAAT |
|
| |
4.2.1.0.1 |
NF-YA (CP1A, CBF-B) |
|
 |
T09472 |
 |
 |
 |
| |
4.2.1.0.1.1 |
NF-YA, isoform 1 (long) |
|
 |
T00615 |
|
| |
4.2.1.0.1.2 |
NF-YA, isoform 2 (short) |
|
 |
T09471 |
|
| |
4.2.1.0.2 |
NF-YB (CP1B, CBF-A) |
|
 |
T00616 |
 |
 |
 |
| |
4.2.1.0.3 |
NF-YC |
|
 |
T02477 |
 |
 |
 |
|
| 5 |
Superclass: α-Helices exposed by β-structures |
 |
| |
5.1 |
Class: MADS box factors |
 |
 |
| |
5.1.1 |
Family: Regulators of differentiation |
CTAAAAATAG |
|
| |
5.1.1.1 |
Subfamily: MEF-2 |
CTAAAAATAG |
|
| |
5.1.1.1.1 |
MEF-2A |
 |
 |
T15781 |
 |
 |
 |
| |
5.1.1.1.1.1 |
MEF-2A, isoform 1 (MEF2) |
|
 |
T00505 |
|
| |
5.1.1.1.1.2 |
MEF-2A, isoform 2 (aMEF2) |
|
 |
T06051 |
|
| |
5.1.1.1.1.3 |
MEF-2A, isoform 3 (RSRFC4) |
|
 |
T11401 |
|
| |
5.1.1.1.2 |
MEF-2B (RSRFR2, xMEF2) |
 |
 |
T11282 |
 |
 |
 |
| |
5.1.1.1.2.1 |
MEF-2B, isoform 1 |
|
 |
T01785 |
|
| |
5.1.1.1.2.2 |
MEF-2B, isoform 2 |
|
 |
T02501 |
|
| |
5.1.1.1.2.3 |
MEF-2B, isoform 3 |
|
 |
T02502 |
|
| |
5.1.1.1.2.4 |
MEF-2B, isoform 4 |
|
 |
T02503 |
|
| |
5.1.1.1.3 |
MEF-2C |
|
 |
T09547 |
 |
 |
|
| |
5.1.1.1.3.1 |
MEF-2C, isoform 1 |
|
 |
T09397 |
|
| |
5.1.1.1.3.2 |
MEF-2C, isoform 2 |
|
 |
T01769 |
|
| |
5.1.1.1.3.3 |
MEF-2C, isoform 3 |
|
 |
T09399 |
|
| |
5.1.1.1.3.4 |
MEF-2C, isoform 4 |
|
 |
T09400 |
|
| |
5.1.1.1.3.5 |
MEF-2C, isoform 5 |
|
 |
T06566 |
|
| |
5.1.1.1.4 |
MEF-2D |
 |
 |
T17720 |
 |
 |
|
| |
5.1.1.1.4.1 |
MEF-2D, isoform 1 (non-muscle) |
|
 |
T02506 |
|
| |
5.1.1.1.4.2 |
MEF-2D, isoform 2 (muscle) |
|
 |
T02505 |
|
| |
5.1.2 |
Family: Responders to external signals (SRF/RLM1) |
CCATATATGG |
|
| |
5.1.2.0.1 |
SRF |
 |
 |
T05114 |
 |
 |
 |
| |
5.2 |
Class: E2-related factors |
|
| |
5.2.1 |
Family: E2 |
|
|
| |
5.3 |
Class: SAND domain factors |
 |
 |
| |
5.3.1 |
Family: AIRE |
ATTGGTTATATTGGTTA |
|
| |
5.3.1.0.1 |
AIRE |
|
 |
T05991 |
 |
 |
|
| |
5.3.1.0.1.1 |
AIRE-1, isoform 1a |
|
 |
T11050 |
|
| |
5.3.1.0.1.2 |
AIRE-2, isoform 1b |
|
 |
T18752 |
|
| |
5.3.1.0.1.3 |
AIRE-3, isoform 1c |
|
 |
T18753 |
|
| |
5.3.1.0.1.4 |
AIRE-3, isoform 1d |
|
 |
T18757 |
|
| |
5.3.1.0.1.5 |
AIRE-1, isoform 2a |
|
 |
T18758 |
|
| |
5.3.1.0.1.6 |
AIRE-2, isoform 2b |
|
 |
T18759 |
|
| |
5.3.1.0.1.7 |
AIRE-3, isoform 2c |
|
 |
T18760 |
|
| |
5.3.1.0.1.8 |
AIRE-3, isoform 2d |
|
 |
T18761 |
|
| |
5.3.1.0.1.9 |
AIRE-1, isoform 3a |
|
 |
T18754 |
|
| |
5.3.1.0.1.10 |
AIRE-2, isoform 3b |
|
 |
T18755 |
|
| |
5.3.1.0.1.11 |
AIRE-3, isoform 3c |
|
 |
T18762 |
|
| |
5.3.1.0.1.12 |
AIRE-3, isoform 3d |
|
 |
T18756 |
|
| |
5.3.2 |
Family: DEAF |
TTTCCG |
|
| |
5.3.2.0.1 |
DEAF1 |
|
 |
T14829 |
 |
 |
|
| |
5.3.2.0.1.1 |
DEAF1, isoform 1 (DF1) |
|
 |
T14830 |
|
| |
5.3.2.0.1.2 |
DEAF1, isoform 2 (DF1-VAR1) |
|
 |
T26083 |
|
| |
5.3.3 |
Family: GMEB |
CTTCTGTATGAGCGCCAGTAT |
|
| |
5.3.3.1 |
Subfamily: GMEB |
|
|
| |
5.3.3.1.1 |
GMEB-1 |
|
 |
T05070 |
 |
 |
 |
| |
5.3.3.1.2 |
GMEB-2 |
 |
 |
T18959 |
 |
 |
|
| |
5.3.4 |
Family: Sp110 |
|
|
| |
5.3.4.0.1 |
Sp110 |
|
 |
T20126 |
 |
 |
 |
| |
5.3.5 |
Family: Sp140/Sp100 |
|
|
| |
5.3.5.1 |
Subfamily: Sp140-like factors |
|
|
| |
5.3.5.1.1 |
Sp140 |
|
 |
T |
 |
 |
|
| |
5.3.5.2 |
Subfamily: Sp100 |
|
|
| |
5.3.5.2.1 |
Sp100 |
|
 |
T16749 |
 |
 |
 |
| |
5.3.5.2.1.1 |
Sp100, isoform 1 |
|
 |
T16748 |
|
| |
5.3.5.2.1.2 |
Sp100, isoform 2 |
|
 |
T16750 |
|
|
| 6 |
Superclass: Immunoglobulin fold |
 |
| |
6.1 |
Class: Rel homology region (RHR) factors |
 |
 |
| |
6.1.1 |
Family: NF-kappaB-related factors |
|
|
| |
6.1.1.1 |
Subfamily: NF-kappaB p50 subunit-like factors |
GGGAATTTCC |
|
| |
6.1.1.1.1 |
NF-κB p105 (NFKB1) |
 |
 |
T09437 |
 |
 |
 |
| |
6.1.1.1.1.1 |
NF-κB, isoform 1 (p105) |
|
 |
T01924 |
|
| |
6.1.1.1.1.2 |
NF-κB, isoform 1 (p50) |
|
 |
T17574 |
|
| |
6.1.1.1.1.3 |
NF-κB, isoform 2 (p84) |
|
 |
T11413 |
|
| |
6.1.1.1.1.4 |
NF-κB, isoform 3 (p98) |
|
 |
T11415 |
|
| |
6.1.1.1.1.5 |
NF-κB, isoform 4 |
|
 |
T11418 |
|
| |
6.1.1.1.1.6 |
NF-κB, isoform 5 (p70, IκB-γ) |
|
 |
T01940 |
|
| |
6.1.1.1.1.7 |
NF-κB, isoform 6 (p63, IκB-γ-1) |
|
 |
T01941 |
|
| |
6.1.1.1.1.8 |
NF-κB, isoform 7 (p55, IκB-γ-2) |
|
 |
T01942 |
|
| |
6.1.1.1.2 |
NF-κB p100 (NFKB2) |
 |
 |
T19129 |
 |
 |
 |
| |
6.1.1.1.2.1 |
NF-κB, isoform 1 (p100) |
|
 |
T08984 |
|
| |
6.1.1.1.2.2 |
NF-κB, isoform 1 (p52) |
|
 |
T19130 |
|
| |
6.1.1.2 |
Subfamily: NF-kappaB p65 subunit-like factors |
GAAAATTTCC |
|
| |
6.1.1.2.1 |
NF-κB p65 (RelA) |
|
 |
T00595 |
 |
 |
 |
| |
6.1.1.2.1.1 |
NF-κB, isoform 1 (p65) |
|
 |
T09106 |
|
| |
6.1.1.2.1.2 |
NF-κB, isoform 3 (p65Δ) |
|
 |
T09631 |
|
| |
6.1.1.2.2 |
RelB (I-Rel) |
|
 |
T01932 |
 |
 |
 |
| |
6.1.1.2.3 |
c-Rel |
|
 |
T00169 |
 |
 |
 |
| |
6.1.2 |
Family: Ankyrin domain-only factors |
|
|
| |
6.1.2.1 |
Subfamily: IκB-related factors |
|
|
| |
6.1.2.1.1 |
IκBα |
|
 |
T |
 |
 |
|
| |
6.1.2.1.2 |
IκBβ |
|
 |
T00971 |
 |
 |
|
| |
6.1.2.1.3 |
IκBδ |
|
 |
T |
 |
 |
|
| |
6.1.2.1.4 |
IκBε |
|
 |
T22412 |
 |
 |
|
| |
6.1.2.1.5 |
IκBζ (MAIL) |
|
 |
T |
 |
 |
|
| |
6.1.2.1.5.1 |
IκBζ, isoform 1 (MAIL-L) |
|
 |
T |
|
| |
6.1.2.1.5.2 |
IκBζ, isoform 2 (MAIL-S) |
|
 |
T |
|
| |
6.1.2.1.5.3 |
IκBζ, isoform 3 (D) |
|
 |
T |
|
| |
6.1.2.1.6 |
IκBR (Tonsl) |
|
 |
T |
 |
 |
|
| |
6.1.2.1.7 |
IκBL |
|
 |
T |
 |
 |
|
| |
6.1.2.1.8 |
BCL-3 |
|
 |
T |
 |
 |
|
| |
6.1.2.2 |
Subfamily: RelA-associated inhibitor |
|
|
| |
6.1.2.2.1 |
RAI (iASPP) |
|
 |
T27841 |
 |
 |
|
| |
6.1.3 |
Family: NFAT-related factors |
ATGGAAA |
|
| |
6.1.3.0.1 |
NFATc1 |
 |
 |
T01947 |
 |
 |
 |
| |
6.1.3.0.1.1 |
NFATc1, isoform α |
|
 |
T08999 |
|
| |
6.1.3.0.1.2 |
NFATc1, isoform α' |
|
 |
|
|
| |
6.1.3.0.1.3 |
NFATc1, isoform β |
|
 |
T17544 |
|
| |
6.1.3.0.2 |
NFATc2 (NFATp, NFAT1) |
|
 |
T15616 |
 |
 |
 |
| |
6.1.3.0.2.1 |
NFATc2, isoform A |
|
 |
T01944 |
|
| |
6.1.3.0.2.2 |
NFATc2, isoform B |
|
 |
T19124 |
|
| |
6.1.3.0.2.3 |
NFATc2, isoform C |
|
 |
T19123 |
|
| |
6.1.3.0.2.4 |
NFATc2, isoform D |
|
 |
T15617 |
|
| |
6.1.3.0.3 |
NFATc3 (NFATx, NFAT4) |
|
 |
T01949 |
 |
 |
|
| |
6.1.3.0.3.1 |
NFATc3, isoform 1 (X1) |
|
 |
|
|
| |
6.1.3.0.3.2 |
NFATc3, isoform 2 (X2) |
|
 |
|
|
| |
6.1.3.0.3.3 |
NFATc3, isoform 3 (ΔX) |
|
 |
|
|
| |
6.1.3.0.4 |
NFATc4 (NFAT3) |
|
 |
T05523 |
 |
 |
|
| |
6.1.3.0.4.1 |
NFATc4, isoform 1 (I-IXL) |
|
 |
T10410 |
|
| |
6.1.3.0.4.2 |
NFATc4, isoform 2 (I-IXi) |
|
 |
T26208 |
|
| |
6.1.3.0.5 |
NFAT5 (TonEBP) |
 |
 |
T10408 |
 |
 |
 |
| |
6.1.3.0.5.1 |
NFAT5, isoform 1 (C) |
|
 |
T10407 |
|
| |
6.1.3.0.5.2 |
NFAT5, isoform 2 (D) |
|
 |
T30305 |
|
| |
6.1.3.0.5.3 |
NFAT5, isoform 3 (A) |
|
 |
T30306 |
|
| |
6.1.3.0.5.4 |
NFAT5, isoform 4 |
|
 |
T30307 |
|
| |
6.1.4 |
Family: CSL-related factors |
CGTGGGAA |
|
| |
6.1.4.1 |
Subfamily: M |
CGTGGGAA |
|
| |
6.1.4.1.1 |
RBP-Jκ |
|
 |
T00440 |
 |
 F F |
 |
| |
6.1.4.1.1.1 |
RBP-Jκ, isoform 1 |
|
 |
T09441 |
|
| |
6.1.4.1.1.2 |
RBP-Jκ, isoform 2 |
|
 |
T26313 |
|
| |
6.1.4.1.2 |
RBP-JL |
|
 |
T17276 |
 |
 |
|
| |
6.1.5 |
Family: Early B-Cell Factor-related factors |
TCCCTAGGGA |
|
| |
6.1.5.0.1 |
EBF1 (COE1) |
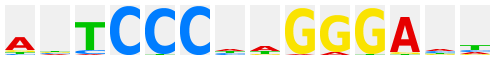 |
 |
T09926 |
 |
 |
|
| |
6.1.5.0.1.1 |
EBF1, isoform 1 (long, 8) |
|
 |
T01112 |
|
| |
6.1.5.0.1.2 |
EBF1, isoform 2 (short, O) |
|
 |
T18848 |
|
| |
6.1.5.0.2 |
EBF2 (COE2) |
|
 |
T05006 |
 |
 |
|
| |
6.1.5.0.3 |
EBF3 (COE3) |
|
 |
T05008 |
 |
 |
|
| |
6.1.5.0.3.1 |
EBF3, isoform long (9L) |
|
 |
T14704 |
|
| |
6.1.5.0.3.2 |
EBF3, isoform short (0S) |
|
 |
T15857 |
|
| |
6.1.5.0.4 |
EBF4 (COE4) |
|
 |
T19885 |
 |
 F F |
|
| |
6.1.5.0.4.1 |
EBF4, isoform 1 (4-11) |
|
 |
T19886 |
|
| |
6.1.5.0.4.2 |
EBF4, isoform 2 (4-14) |
|
 |
T19887 |
|
| |
6.1.5.0.4.3 |
EBF4, isoform 3 (4-23) |
|
 |
T19884 |
|
| |
6.1.5.0.4.4 |
EBF4, isoform 4 (4-132) |
|
 |
T19888 |
|
| |
6.1.5.0.4.5 |
EBF4, isoform 5 (4S) |
|
 |
T19889 |
|
| |
6.2 |
Class: STAT domain factors |
 |
 |
| |
6.2.1 |
Family: STAT factors |
TTCCCGGAA |
|
| |
6.2.1.0.1 |
STAT1 |
|
 |
T08951 |
 |
 |
 |
| |
6.2.1.0.2 |
STAT2 |
|
 |
T11001 |
 |
 |
|
| |
6.2.1.0.3 |
STAT3 |
|
 |
T01574 |
 |
 |
|
| |
6.2.1.0.3.1 |
STAT3, isoform A |
|
 |
T08657 |
|
| |
6.2.1.0.3.2 |
STAT3, isoform B |
|
 |
T08658 |
|
| |
6.2.1.0.3.3 |
STAT3, isoform Del-701 |
|
 |
T19278 |
|
| |
6.2.1.0.4 |
STAT4 |
|
 |
T01576 |
 |
 |
|
| |
6.2.1.0.5 |
STAT5A |
|
 |
T01579 |
 |
 |
|
| |
6.2.1.0.6 |
STAT5B |
|
 |
T00944 |
 |
 |
|
| |
6.2.1.0.7 |
STAT6 |
|
 |
T01581 |
 |
 |
|
| |
6.3 |
Class: p53 domain factors |
 |
 |
| |
6.3.1 |
Family: p53-related factors |
GGACATGCCCGGGCATGTCC |
|
| |
6.3.1.0.1 |
p53 |
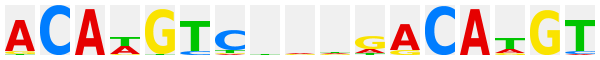 |
 |
T01806 |
 |
 |
 |
| |
6.3.1.0.2 |
p63 |
 |
 |
T06293 |
 |
 |
 |
| |
6.3.1.0.2.1 |
p63, isoform 1 (p63α) |
|
 |
T06168 |
|
| |
6.3.1.0.2.2 |
p63, isoform 2 (ΔN-p63α) |
|
 |
T06011 |
|
| |
6.3.1.0.2.3 |
p63, isoform 3 (p63β) |
|
 |
T06290 |
|
| |
6.3.1.0.2.4 |
p63, isoform 4 (ΔN-p63β) |
|
 |
T06010 |
|
| |
6.3.1.0.2.5 |
p63, isoform 5 (p63γ) |
|
 |
T06291 |
|
| |
6.3.1.0.2.6 |
p63, isoform 6 (ΔN-p63γ) |
|
 |
T06292 |
|
| |
6.3.1.0.3 |
p73 |
 |
 |
T06287 |
 |
 |
 |
| |
6.3.1.0.3.1 |
p73, isoform 1 (α) |
|
 |
T06008 |
|
| |
6.3.1.0.3.2 |
p73, isoform 2 (dN-α) |
|
 |
T06288 |
|
| |
6.3.1.0.3.3 |
p73, isoform 3 |
|
 |
T26229 |
|
| |
6.3.1.0.3.4 |
p73, isoform 4 |
|
 |
T26230 |
|
| |
6.4 |
Class: Runt domain factors |
 |
 |
| |
6.4.1 |
Family: Runt-related factors |
TGTGGT |
|
| |
6.4.1.0.1 |
Runx2 (PEBP2αA, CBF-α1) |
 |
 |
T08443 |
 |
 F F |
|
| |
6.4.1.0.1.1 |
Runx2, isoform 1 |
|
 |
T09535 |
|
| |
6.4.1.0.1.2 |
Runx2, isoform 2 |
|
 |
T02266 |
|
| |
6.4.1.0.1.3 |
Runx2, isoform 3 (PEBP2αA1) |
|
 |
T01062 |
|
| |
6.4.1.0.1.4 |
Runx2, isoform 4 (PEBP2αA2) |
|
 |
T01063 |
|
| |
6.4.1.0.1.5 |
Runx2, isoform 5 (G1) |
|
 |
T02318 |
|
| |
6.4.1.0.1.6 |
Runx2, isoform 6 (G2) |
|
 |
T09536 |
|
| |
6.4.1.0.1.7 |
Runx2, isoform 7 (U1) |
|
 |
T02319 |
|
| |
6.4.1.0.1.8 |
Runx2, isoform 8 (Y1) |
|
 |
T02320 |
|
| |
6.4.1.0.1.9 |
Runx2, isoform 9 (Y2) |
|
 |
T09537 |
|
| |
6.4.1.0.2 |
Runx1 (PEBP2αB, CBF-α2, AML-1) |
|
 |
T06598 |
 |
 |
 |
| |
6.4.1.0.2.1 |
Runx1, isoform 1 (AML-1B, PEBP2αB1) |
|
 |
T02235 |
|
| |
6.4.1.0.2.2 |
Runx1, isoform 2 (AML-1A, PEBP2αB2) |
|
 |
T02241 |
|
| |
6.4.1.0.2.3 |
Runx1, isoform 3 |
|
 |
T17394 |
|
| |
6.4.1.0.2.4 |
Runx1, isoform 4 (AML-1C) |
|
 |
T17393 |
|
| |
6.4.1.0.2.5 |
Runx1, isoform 5 |
|
 |
T17386 |
|
| |
6.4.1.0.3 |
Runx3 (PEBP2αC, CBF-α3, AML-2) |
 |
 |
T05409 |
 |
 |
|
| |
6.4.1.0.3.1 |
Runx3, isoform 1 (AML-2α) |
|
 |
T19218 |
|
| |
6.4.1.0.3.2 |
Runx3, isoform 2 (AML-2β) |
|
 |
T09273 |
|
| |
6.5 |
Class: T-Box factors |
 |
 |
| |
6.5.1 |
Family: Brachyury-related factors |
TCACACCTAGGTGTGAAATT |
|
| |
6.5.1.0.1 |
T (Brachyury) |
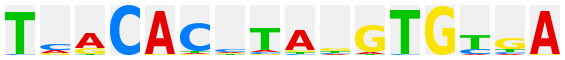 |
 |
T01526 |
 |
 |
 |
| |
6.5.1.0.2 |
TBX19 |
 |
 |
T18412 |
 |
 |
|
| |
6.5.2 |
Family: TBrain-related factors |
AGGTGTGAA |
|
| |
6.5.2.0.1 |
TBR-1 |
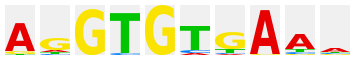 |
 |
T21429 |
 |
 |
|
| |
6.5.2.0.2 |
TBR-2 (EOMES) |
 |
 |
T16915 |
 |
 F F |
|
| |
6.5.2.0.2.1 |
TBR-2, isoform 1 |
|
 |
T16857 |
|
| |
6.5.2.0.2.2 |
TBR-2, isoform 2 |
|
 |
T26098 |
|
| |
6.5.2.0.3 |
TBX21 (T-bet) |
 |
 |
T19296 |
 |
 |
|
| |
6.5.3 |
Family: TBX1-related factors |
|
|
| |
6.5.3.0.1 |
TBX1 |
 |
 |
T04405 |
 |
 |
 |
| |
6.5.3.0.1.1 |
TBX1, isoform 1 |
|
 |
T11341 |
|
| |
6.5.3.0.1.2 |
TBX1, isoform 2 |
|
 |
T26329 |
|
| |
6.5.3.0.2 |
TBX10 (TBX7) |
|
 |
T19295 |
 |
 |
|
| |
6.5.3.0.2.1 |
TBX10, isoform 1 |
|
 |
T19294 |
|
| |
6.5.3.0.2.2 |
TBX10, isoform 2 |
|
 |
T19293 |
|
| |
6.5.3.0.3 |
TBX15 (TBX14) |
 |
 |
T16162 |
 |
 |
|
| |
6.5.3.0.4 |
TBX18 |
|
 |
T04418 |
 |
 |
|
| |
6.5.3.0.5 |
TBX20 |
 |
 |
T04424 |
 |
 |
|
| |
6.5.3.0.5.1 |
TBX20, isoform 1 |
|
 |
T11346 |
|
| |
6.5.3.0.5.2 |
TBX20, isoform 2 |
|
 |
T26330 |
|
| |
6.5.3.0.6 |
TBX22 |
|
 |
T16745 |
 |
 |
|
| |
6.5.4 |
Family: TBX2-related factors |
AGGTGTGAG |
|
| |
6.5.4.0.1 |
TBX2 |
 |
 |
T04358 |
 |
 |
|
| |
6.5.4.0.2 |
TBX3 |
|
 |
T04359 |
 |
 |
 |
| |
6.5.4.0.2.1 |
TBX3, isoform 1 |
|
 |
T11348 |
|
| |
6.5.4.0.2.2 |
TBX3, isoform 2 |
|
 |
T26331 |
|
| |
6.5.4.0.3 |
TBX4 |
 |
 |
T04350 |
 |
 |
|
| |
6.5.4.0.4 |
TBX5 |
 |
 |
T04347 |
 |
 |
 |
| |
6.5.5 |
Family: TBX6-related factors |
AATTTCACACCTAGGTGTGAAATT |
|
| |
6.5.5.0.1 |
TBX6 |
|
 |
T04417 |
 |
 |
|
| |
6.5.5.0.1.1 |
TBX6, isoform 1 |
|
 |
T14662 |
|
| |
6.5.5.0.1.2 |
TBX6, isoform 2 |
|
 |
T26490 |
|
| |
6.5.5.0.2 |
MGA (MAD5) = 1.2.6.7.4 |
 |
 |
T03940 |
 |
 |
|
| |
6.5.5.0.2.1 |
Mad5, isoform 1 |
|
 |
T14744 |
|
| |
6.5.5.0.2.2 |
Mad5, isoform 2 |
|
 |
T26191 |
|
| |
6.5.5.0.2.3 |
Mad5, isoform 3 |
|
 |
T26192 |
|
| |
6.5.5.0.2.4 |
Mad5, isoform 4 |
|
 |
T26193 |
|
| |
6.6 |
Class: NDT80 domain factors |
 |
| |
6.6.1 |
Family: Myelin gene regulatory factor-related factors |
|
|
| |
6.6.1.0.1 |
Myelin gene regulatory factor (Myrf, MRF, C11orf9) |
|
 |
T25539 |
 |
 |
 |
| |
6.6.1.0.1.1 |
Myrf, isoform 1 |
|
 |
T25986 |
|
| |
6.6.1.0.1.2 |
Myrf, isoform 2 |
|
 |
T25987 |
|
| |
6.6.1.0.1.3 |
Myrf, isoform 3 |
|
 |
T30300 |
|
| |
6.6.1.0.1.4 |
Myrf, isoform 4 |
|
 |
T30301 |
|
| |
6.7 |
Class: Grainyhead domain factors |
 |
| |
6.7.1 |
Family: Grainyhead-related factors |
|
|
| |
6.7.1.1 |
Subfamily: GRH-like proteins |
|
|
| |
6.7.1.1.1 |
GRH-L1 (CP2-L2, LBP-32, MGR) |
 |
 |
T19962 |
 |
 F F |
|
| |
6.7.1.1.1.1 |
p70 MGR, isoform 1 |
|
 |
T19961 |
|
| |
6.7.1.1.1.2 |
p49 MGR, isoform 2 |
|
 |
T19963 |
|
| |
6.7.1.1.2 |
GRH-L2 (CP2-L3) |
|
 |
T19968 |
 |
 |
|
| |
6.7.1.1.3 |
GRH-L3 (CP2-L4) |
|
 |
T19975 |
 |
 |
|
| |
6.7.2 |
Family: CP2-related factors |
GCACAAACCAG |
|
| |
6.7.2.0.1 |
CP2 (LSF, SEF) |
 |
 |
T00152 |
 |
 |
|
| |
6.7.2.0.1.1 |
CP2, isoform 1 |
|
 |
T08971 |
|
| |
6.7.2.0.1.2 |
CP2, isoform 2 |
|
 |
T05680 |
|
| |
6.7.2.0.1.3 |
CP2, isoform 3 |
|
 |
T19332 |
|
| |
6.7.2.0.2 |
CP2-L1 (LBP-9, CRTR-1) |
|
 |
T05733 |
 |
 |
|
| |
6.7.2.0.3 |
UBP-1 (LBP-1, SEF, CP2b) |
|
 |
T01597 |
 |
 |
|
| |
6.7.2.0.3.1 |
UBP-1, isoform 1 |
|
 |
T19355 |
|
| |
6.7.2.0.3.2 |
UBP-1, isoform 2 |
|
 |
T08970 |
|
|
| 7 |
Superclass:
β-Hairpin exposed by an α/β-scaffold |
 |
| |
7.1 |
Class: SMAD/NF-1 DNA-binding domain factors |
 |
 |
| |
7.1.1 |
Family: SMAD factors |
GTCTAGAC |
|
| |
7.1.1.1 |
Subfamily: Regulatory Smads (R-Smad) |
|
|
| |
7.1.1.1.1 |
SMAD1 |
|
 |
T04254 |
 |
 |
 |
| |
7.1.1.1.2 |
SMAD2 |
|
 |
T04273 |
 |
 |
|
| |
7.1.1.1.2.1 |
SMAD2, isoform long |
|
 |
T09086 |
|
| |
7.1.1.1.2.2 |
SMAD2, isoform short |
|
 |
T26297 |
|
| |
7.1.1.1.3 |
SMAD3 |
 |
 |
T04210 |
 |
 |
 |
| |
7.1.1.1.4 |
SMAD5 |
|
 |
T04213 |
 |
 |
|
| |
7.1.1.1.5 |
SMAD9 (SMAD8) |
|
 |
T04215 |
 |
 |
|
| |
7.1.1.2 |
Subfamily: Co-activating Smads (Co-Smad) |
|
|
| |
7.1.1.2.1 |
SMAD4 |
|
 |
T05067 |
 |
 |
 |
| |
7.1.1.3 |
Subfamily: Repressor-Smads (I-Smad) |
|
|
| |
7.1.1.3.1 |
SMAD6 |
|
 |
T04236 |
 |
 |
|
| |
7.1.1.3.2 |
SMAD7 |
|
 |
T04234 |
 |
 |
|
| |
7.1.1.3.2.1 |
SMAD7, isoform A |
|
 |
T08480 |
|
| |
7.1.1.3.2.2 |
SMAD7, isoform B |
|
 |
T25680 |
|
| |
7.1.2 |
Family: Nuclear factor 1 |
TTGGCTATATGCCAA |
|
| |
7.1.2.0.1 |
NF-1A (NF-IA) |
 |
 |
T15519 |
 |
 |
|
| |
7.1.2.0.1.1 |
NF-1A, isoform 1 |
|
 |
T26210 |
|
| |
7.1.2.0.1.2 |
NF-1A, isoform 2 |
|
 |
T19125 |
|
| |
7.1.2.0.1.3 |
NF-1A, isoform 3 |
|
 |
T19126 |
|
| |
7.1.2.0.1.4 |
NF-1A, isoform 4 |
|
 |
T26211 |
|
| |
7.1.2.0.1.5 |
NF-1A, isoform 5 |
|
 |
T08177 |
|
| |
7.1.2.0.1.6 |
NF-1A, isoform 6 |
|
 |
T08175 |
|
| |
7.1.2.0.1.7 |
NF-1A, isoform 7 |
|
 |
T08176 |
|
| |
7.1.2.0.2 |
NF-1B |
 |
 |
T09201 |
 |
 |
|
| |
7.1.2.0.2.1 |
NF-1B, isoform 1 |
|
 |
T08170 |
|
| |
7.1.2.0.2.2 |
NF-1B, isoform 2 |
|
 |
T01909 |
|
| |
7.1.2.0.2.3 |
NF-1B, isoform 3 |
|
 |
T01910 |
|
| |
7.1.2.0.3 |
NF-1C |
|
 |
T09202 |
 |
 |
|
| |
7.1.2.0.3.1 |
NF-1C, isoform 1 (C1A, C2) |
|
 |
T08183 |
|
| |
7.1.2.0.3.2 |
NF-1C, isoform 2 (C1B) |
|
 |
T08184 |
|
| |
7.1.2.0.3.3 |
NF-1C, isoform 3 (C5) |
|
 |
T08188 |
|
| |
7.1.2.0.3.4 |
NF-1C, isoform 4 (C8) |
|
 |
T08189 |
|
| |
7.1.2.0.3.5 |
NF-1C, isoform 5 (C9) |
|
 |
T08190 |
|
| |
7.1.2.0.3.6 |
NF-1C, isoform 6 (C10) |
|
 |
T08185 |
|
| |
7.1.2.0.3.7 |
NF-1C, isoform 7 (C11) |
|
 |
T08186 |
|
| |
7.1.2.0.4 |
NF-1X |
 |
 |
T09203 |
 |
 |
|
| |
7.1.2.0.4.1 |
NF-1X, isoform 1 |
|
 |
T08173 |
|
| |
7.1.2.0.4.2 |
NF-1X, isoform 2 |
|
 |
T08174 |
|
| |
7.1.2.0.4.3 |
NF-1X, isoform 3 |
|
 |
T08194 |
|
| |
7.2 |
Class: GCM domain factors |
 |
| |
7.2.1 |
Family: GCM factors |
|
|
| |
7.2.1.0.1 |
GCMa (GCM1) |
 |
 |
T02307 |
 |
 |
 |
| |
7.2.1.0.2 |
GCMb (GCM2) |
 |
 |
T02308 |
 |
 |
|
|
| 8 |
Superclass:
β-Sheet binding to DNA |
 |
| |
8.1 |
Class: TATA-binding proteins |
 |
 |
| |
8.1.1 |
Family: TBP-related factors |
TATAAA) |
|
| |
8.1.1.0.1 |
TBP |
|
 |
T00796 |
 |
 |
 |
| |
8.1.1.0.2 |
TBP-L1 (TRF2) |
|
 |
T23344 |
 |
 |
|
| |
8.1.1.0.3 |
TBP-L2 (TRF3) |
|
 |
T20141 |
 |
 |
|
| |
8.2 |
Class: A.T hook factors |
 |
 |
| |
8.2.1 |
Family: HMGA factors |
GGAAATT |
|
| |
8.2.1.0.1 |
HMGA1 (HMGI(Y)) |
|
 |
T01852 |
 |
 |
 |
| |
8.2.1.0.1.1 |
HMGA1a, isoform HMG-I |
|
 |
T19006 |
|
| |
8.2.1.0.1.2 |
HMGA1b, isoform HMG-Y |
|
 |
T10672 |
|
| |
8.2.1.0.2 |
HMGA2 (HMGI-C) |
|
 |
T01860 |
 |
 |
|
|
| 9 |
Superclass: β-Barrel DNA-binding domains |
 |
| |
9.1 |
Class: Cold-shock domain factors |
 |
 |
| |
9.1.1 |
Family: Dbp factors |
CCAATCAG |
|
| |
9.1.1.1 |
Subfamily: A (DbpA-like) |
|
|
| |
9.1.1.1.1 |
DbpA (YBX3, CSDA) |
|
 |
T11073 |
 |
 |
|
| |
9.1.1.1.1.1 |
DbpA, isoform 1 |
|
 |
|
|
| |
9.1.1.1.1.2 |
DbpA, isoform 2 |
|
 |
T26082 |
|
| |
9.1.1.2 |
Subfamily: B (DbpB/YB-1-like) |
|
|
| |
9.1.1.2.1 |
YB-1 (DbpB, YBX1) |
|
 |
T02496 |
 |
 |
 |
| |
9.1.1.3 |
Subfamily: C (FRG Y2-like) |
|
|
| |
9.1.1.3.1 |
YBX2 (DbpC, MSY2, FRG Y2) |
|
 |
T |
 |
 |
|
| |
9.1.1.3.1.1 |
YBX2, isoform 1 |
|
 |
T |
|
| |
9.1.1.3.1.2 |
YBX2, isoform 2 |
|
 |
T |
|
|
| 0 |
Superclass: Yet undefined DNA-binding domains |
 |
| |
0.1 |
Class: AXUD/CSRNP domain factors |
 |
| |
0.1.1 |
Family: CSRNP factors |
AGAGTG |
|
| |
0.1.1.0.1 |
CSRNP-1 (AXUD1, TAIP3) |
|
 |
T |
 |
 |
|
| |
0.1.1.0.2 |
CSRNP-2 (TAIP12) |
|
 |
T |
 |
 |
|
| |
0.1.1.0.3 |
CSRNP-3 (TAIP2) |
|
 |
T27840 |
 |
 F F |
|
| |
0.1.1.0.3.1 |
CSRNP-3, isoform 1 |
|
 |
T27918 |
|
| |
0.1.1.0.3.2 |
CSRNP-3, isoform 2 |
|
 |
T27343 |
|
| |
0.2 |
Class: NonO domain factors |
 |
| |
0.2.1 |
Family: NonO-related factors |
|
|
| |
0.2.1.0.1 |
NONO (NMT55, p54(nrb), NRB54) |
|
 |
T26600 |
 |
 |
 |
| |
0.2.1.0.1.1 |
NONO, isoform 1 |
|
 |
T26833 |
|
| |
0.2.1.0.1.2 |
NONO, isoform 2 |
|
 |
T26845 |
|
| |
0.2.1.0.2 |
SFPQ (PSF) |
|
 |
T |
 |
 |
|
| |
0.2.1.0.3 |
PSPC1 (PSP1, Paraspeckle component 1) |
|
 |
T27851 |
 |
 |
 |
| |
0.2.1.0.3.1 |
PSPC1, isoform 1 (PSP1-α) |
|
 |
T27356 |
|
| |
0.2.1.0.3.2 |
PSPC1, isoform 2 (PSP1-β) |
|
 |
T27931 |
|
| |
0.3 |
Class: Leucine-rich repeat flightless-interacting proteins |
 |
| |
0.3.1 |
Family: LRRFIP factors |
AGCCCCCGGCG |
|
| |
0.3.1.0.1 |
LRRFIP1 (GCF2, TRIP) |
|
 |
T |
 |
 |
|
| |
0.3.1.0.1.1 |
LRRFIP1, isoform 1 |
|
 |
T |
|
| |
0.3.1.0.1.2 |
LRRFIP1, isoform 2 |
|
 |
T |
|
| |
0.3.1.0.1.3 |
LRRFIP1, isoform 3 |
|
 |
T |
|
| |
0.3.1.0.2 |
LRRFIP2 |
|
 |
T27834 |
 |
 |
|
| |
0.3.1.0.2.1 |
LRRFIP2, isoform 1 |
|
 |
T27888 |
|
| |
0.3.1.0.2.2 |
LRRFIP2, isoform 2 |
|
 |
T27238 |
|
| |
0.4 |
Class: NFX1-type putative zinc finger factors |
 |
| |
0.4.1 |
Family: NFX1 |
CCTAGCAACAGATG |
|
| |
0.4.1.0.1 |
NFX1 (NFX2) |
|
 |
T20048 |
 |
 |
|
| |
0.4.1.0.1.1 |
NFX1, isoform 1 (NFX-123) |
|
 |
T20047 |
|
| |
0.4.1.0.1.2 |
NFX1, isoform 2 |
|
 |
T20049 |
|
| |
0.4.1.0.1.3 |
NFX1, isoform 3 (NFX-91) |
|
 |
T20050 |
|
| |
0.4.1.0.2 |
NFXL1 (OZFP) |
|
 |
T |
 |
 |
|
| |
0.4.1.0.3 |
ZNFX1 |
|
 |
T |
 |
 |
|
| |
0.0 |
Class: Uncharacterized |
 |
| |
0.0.1 |
Family: Nuclear localized protein 1 |
|
|
| |
0.0.1.0.1 |
NULP1 (TCF25) |
|
 |
T |
 |
 |
|
| |
0.0.1.0.1.1 |
NULP1, isoform 1 |
|
 |
T |
|
| |
0.0.1.0.1.2 |
NULP1, isoform 2 |
|
 |
T |
|
| |
0.0.1.0.1.3 |
NULP1, isoform 3 |
|
 |
T |
|
| |
0.0.1.0.1.4 |
NULP1, isoform 4 |
|
 |
T |
|
| |
0.0.1.0.1.5 |
NULP1, isoform 5 |
|
 |
T |
|
| |
0.0.2 |
Family: PHF5 |
|
|
| |
0.0.2.0.1 |
PHF5A (Ini, SF3b14b) |
|
 |
T |
 |
 |
|
| |
0.0.3 |
Family: RFXANK |
CAGTTGCCTAGCAACTA |
|
| |
0.0.3.0.1 |
RFXANK (ANKRA1, RFXB, Tvl-1) |
|
 |
T11076 |
 |
 |
|
| |
0.0.3.0.1.1 |
RFXANK, isoform 1 (long) |
|
 |
T05445 |
|
| |
0.0.3.0.1.2 |
RFXANK, isoform 2 (short) |
|
 |
T05446 |
|
| |
0.0.4 |
Family: RFXAP |
CAGTTGCCTAGCAACTA |
|
| |
0.0.4.0.1 |
RFXAP |
|
 |
T05447 |
 |
 |
 |
| |
0.0.5 |
Family: PUR |
TGGCCAGGG |
|
| |
0.0.5.0.1 |
Pur-α (PURA, PUR1) |
|
 |
T05167 |
 |
 |
|
| |
0.0.5.0.2 |
Pur-β (PURB) |
|
 |
T05172 |
 |
 |
|
| |
0.0.5.0.3 |
Pur-γ (PURG) |
|
 |
T27852 |
 |
 |
|
| |
0.0.5.0.3.1 |
Pur-γ, isoform 1 (PURG-A) |
|
 |
T27357 |
|
| |
0.0.5.0.3.2 |
Pur-γ, isoform 2 (PURG-B) |
|
 |
T27358 |
|
| |
0.0.6 |
Family: NRF |
CGCATGCGCA |
|
| |
0.0.6.0.1 |
NRF-1 (α-pal) |
 |
 |
T05126 |
 |
 |
|
| |
0.0.6.0.1.1 |
NRF-1, isoform 1 (long) |
|
 |
T10598 |
|
| |
0.0.6.0.1.2 |
NRF-1, isoform 2 (short) |
|
 |
T26228 |
|
| |
| |
| |
|
 : TRANSFAC® class descriptions;
: TRANSFAC® class descriptions;  : new class descriptions;
: new class descriptions;  : additional information about the class and its members (see below);
: additional information about the class and its members (see below); : Link to reviewed mouse entries UniProt;
: Link to reviewed mouse entries UniProt;  : Link to unreviewed mouse entries UniProt;
: Link to unreviewed mouse entries UniProt;  : Link to PDB; preferably to a complex of the corresponding DNA-binding domain (DBD) with DNA, provisionally the DBD alone, referring to the human or a mammalian ortholog; for a few excpetions, illustrative structures of fungal homologs are linked.
: Link to PDB; preferably to a complex of the corresponding DNA-binding domain (DBD) with DNA, provisionally the DBD alone, referring to the human or a mammalian ortholog; for a few excpetions, illustrative structures of fungal homologs are linked.













































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































 F
F


















 F
F












































































 F
F























































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































































 ;
;






























































































































































































































































































































































































